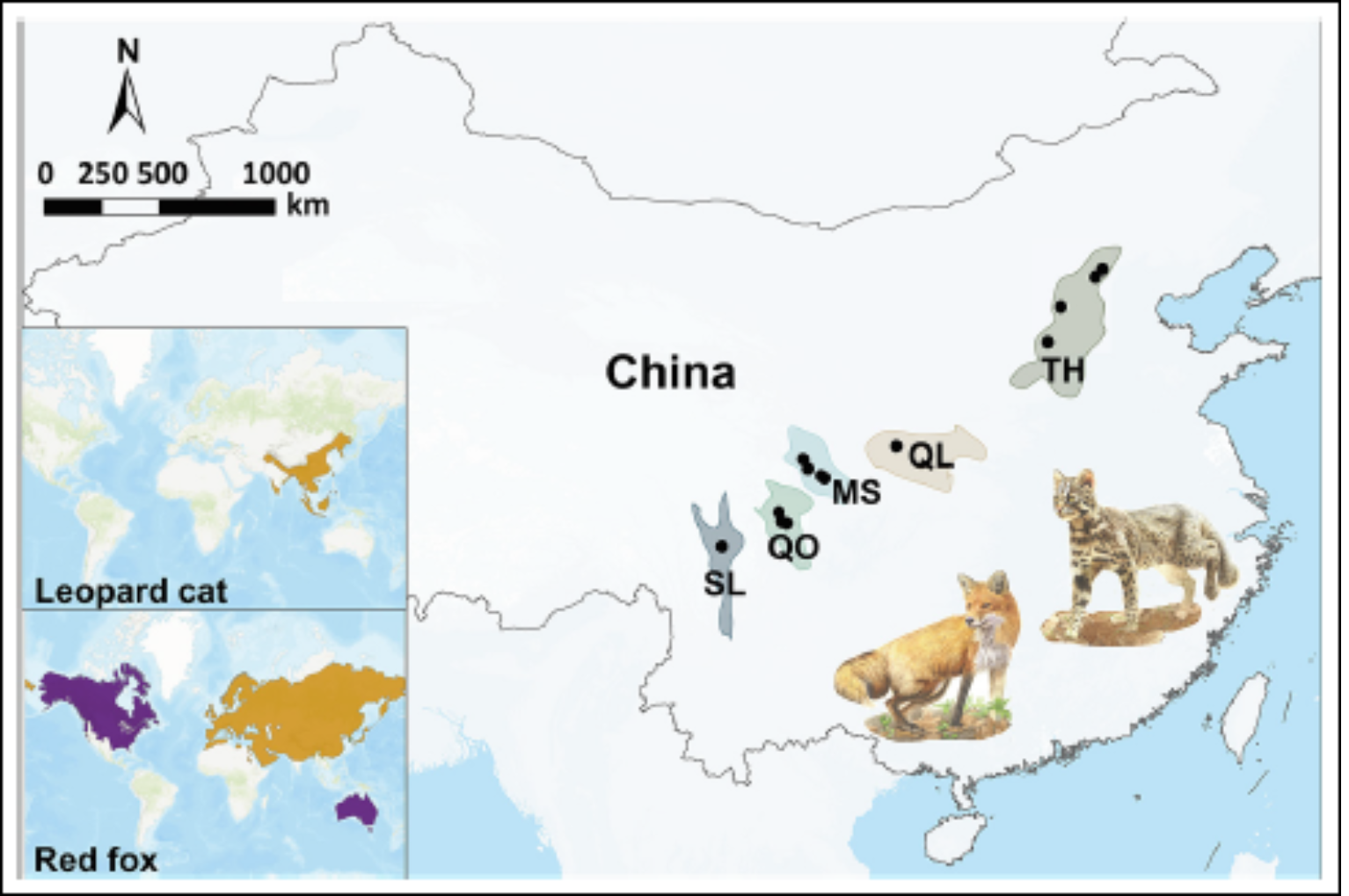
本研究的对象物种(豹猫、赤狐)与样品采集点分布(山脉:TH-太行山, QL-秦岭,MS-岷山,QO-邛崃山, SL-沙鲁里山)

5个山系的豹猫粪便样品中包含的脊椎动物猎物(食物)物种的类群组成
全球生物多样性的快速丧失将严重影响生态系统的功能和服务。为科学且及时地评估区域性生物多样性的现状与动态,亟需高效、灵敏、准确的生物多样性评估技术与手段,以快速查明特定区域或生态系统内的物种组成及其变化,从而确定合理有效的生态保护和管理方案。在陆生脊椎动物中,传统的生物多样性调查方法一般通过捕获和观察的方式,由于其通常具有损伤性,且需要大量的人力物力及专业的形态分类学知识,这种方法越来越不能满足日益增长的区域性和全球性生物多样性评估与保护成效评估的需求。
在陆地生态系统中,中小型食肉动物通常采取机会主义的灵活取食策略,且栖息地适应性强,种群数量较大,粪便样品容易获得。如果我们能够准确高效地辨别其粪便中的食物物种,就能够揭示这些食肉动物所在生物群落的物种组成,它们就可以作为生物多样性“采集器”,提供有关共生物种的生物多样性、种间关系和生态系统动态等方面的重要信息。近年来快速发展的DNA条形码和高通量测序技术为生物多样性调查和监测提供了有力的工具。食肉动物的粪便同时包含捕食者自身及最近取食猎物的DNA,是一种数量丰富且容易收集的生物样本,而标准化的短DNA序列(即条形码)可以准确、灵敏、高效地鉴定捕食者和食物物种。由于中小型食肉动物在世界陆地生态系统内分布广泛,本研究的结果提示,不同地区可以选取本地适宜的“采集器”物种或物种组,以对相应猎物类群的生物多样性进行快速调查。这一新方法将极大地推动传统调查难以开展或形态学鉴定专家缺乏地区的生物多样性本底编目,同时具有快速探测和发现“隐藏”物种及新物种的巨大潜力。

论文第一和第二作者邵昕宁(右)、陆琪(左)在野外采集样品和记录信息

姚蒙(左)、李晟(右)在四川雅江采集粪便样品
北京大学生命科学学院、北京大学生态研究中心姚蒙副研究员、李晟研究员为本论文的共同通讯作者。北京大学生命科学学院博士生邵昕宁为论文的第一作者。该研究得到国家自然科学基金、青藏高原二次科考项目、生态环境部、北京大学及香港海洋公园保育基金的支持。
