正常细胞生长过程中,基因组不稳定主要是来自于DNA复制错误,大约2/3癌症的发生被认为是由于DNA复制错误导致的【Tomasetti & Vogelstein(2015), Science, 347: 78-81;Tomasetti et al. (2017), Science, 355:1330-1334】,而DNA复制错误主要是来自于DNA复制叉的不稳定。DNA复制叉主要由两部分构成(图1):Y字型的DNA结构和DNA复制体(Replisome)。DNA复制体包含一系列与复制相关的蛋白质及蛋白质复合物,其中,最重要的是打开双链DNA模板的CMG(Cdc45-MCM-GINS)解旋酶复合物,以及进行新生链DNA合成的DNA聚合酶——DNAPolα、δ、ε。正常移动的DNA复制叉是相对稳定的,DNA复制体中的CMG解旋酶与DNA聚合酶在物理及生化功能上紧密偶联。但当DNA复制叉遭遇复制障碍停顿下来时,停顿的DNA复制叉被证明是不稳定的,倾向于垮塌。
上个世纪60年代末,研究人员在裂殖酵母里筛选到了一株突变株,命名为rad3-136。很快发现rad3-136突变株对羟基脲【Hydroxyurea (HU), 抑制dNTPs合成,导致DNA复制叉停顿】高度敏感。至此,细胞维持停顿DNA复制叉稳定的机制研究拉开了序幕。Rad3及相关蛋白介导的细胞调控,于1988年被命名为checkpoint调控【Weinert & Hartwell(1988), Science, 241:317-322】, Rad3被归类为ATR激酶。过去50年,前后上千个实验室,用两代人的时间,发表了上万篇研究文章,证明checkpoint是维持停顿DNA复制叉稳定的必需细胞调控。在checkpoint缺失或缺陷的细胞,停顿的DNA复制叉发生垮塌,导致DNA复制不能完成,以及基因组的极度不稳定。但是,研究组在试图阐明checkpoint调控维持DNA复制叉稳定的机制研究方面,碰到了极大阻力,阻力主要是来自于checkpoint调控的靶蛋白极难被确定,已发表文章的结论也互相矛盾,导致checkpoint调控维持DNA复制叉稳定的核心机制长期得不到确定。
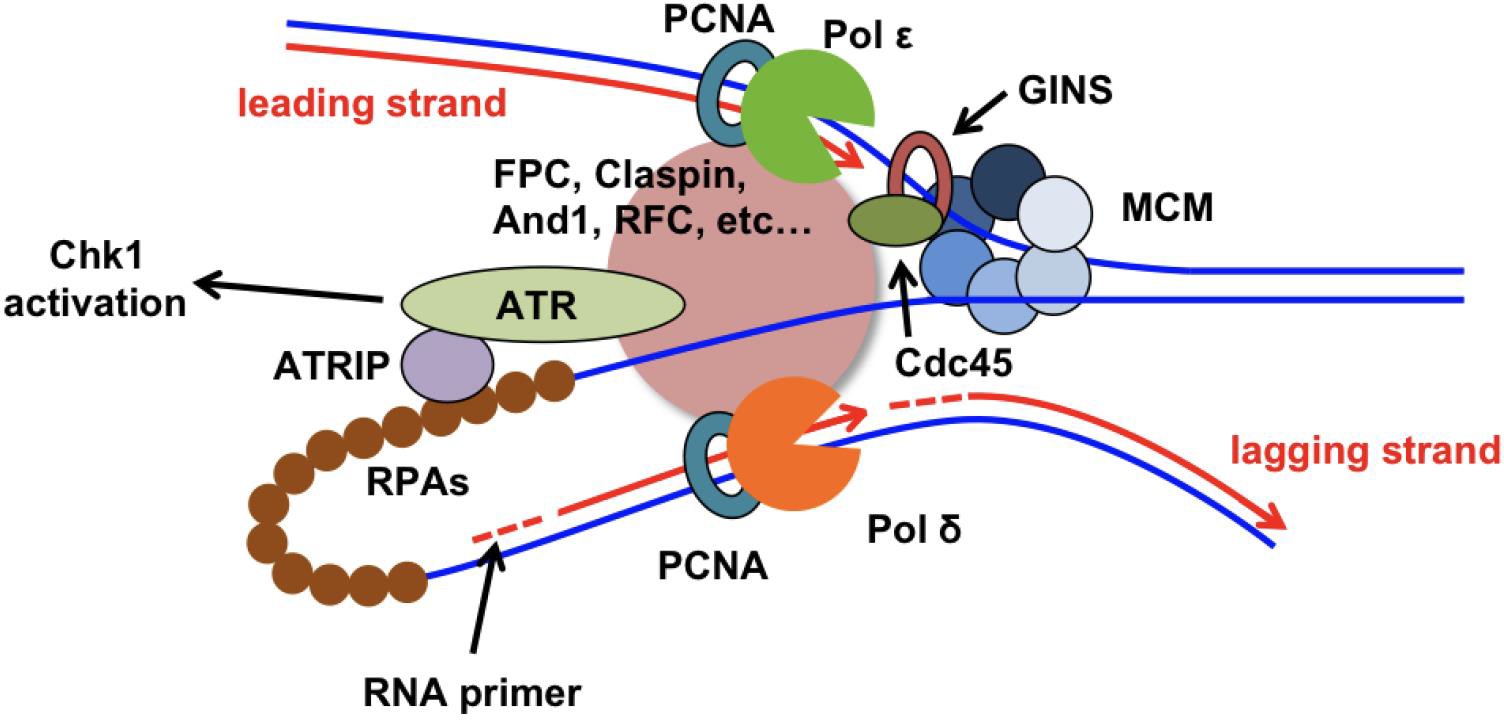
图1 真核细胞DNA复制叉及S期细胞周期检验点【Leman AR, NoguchiE. The replication fork: understanding the eukaryotic replication machinery and the challenges to genome duplication.Genes.2013Mar;4(1):1-32.】。酵母Chk2在高等真核生物的功能同源蛋白是Chk1
通过大规模的遗传筛选,孔道春实验室发现,复制解旋酶CMG(Cdc45-MCM- GINS)复合体的一个亚基Cdc45的突变能极大降低(600-3000 倍)checkpoint通路缺陷的细胞对HU 的敏感性,暗示Cdc45或CMG复合体可能被checkpoint调控。通过一系列遗传及生化分析,证明Rad3ATR-Cds1Chk2 checkpoint 通路直接调控DNA复制解旋酶CMG复合体。孔道春实验室发现:当DNA复制叉停顿后,Cds1Chk2 同时磷酸化Cdc45 亚基上的三个丝氨酸残基(且这三个Chk2/Chk1 磷酸化位点在不同物种中相对保守)。这三个丝氨酸残基的磷酸化导致CMG复制解旋酶活性深度降低。CMG复制解旋酶活性的降低使得它不能脱离停顿复制叉,不能与被阻挡的DNA 聚合酶分开。
这样,停顿的DNA复制体(Replisome)及复制叉的生化完整性得到了维持,从而阻止了停顿DNA 复制叉的垮塌。
为什么停顿复制叉里的复制解旋酶与DNA聚合酶会倾向于分开呢?这关联到两件事情:一是DNA复制体或DNA复制叉的基本生化特性;二是正常细胞生长过程中导致复制叉停顿的基本原因。DNA复制体中,DNA Pol ε负责合成前导链(leading strand)合成, DNA Polα和δ负责后随链(laggingstrand)合成。由于后随链的合成稍微滞后于CMG解旋酶介导的DNA链解开,导致后随链的DNA模板上恒定的出现一段约200核苷酸长度的单链DNA区域。如果该单链DNA区域有形成DNA二级结构的序列,如G4、三股链、发夹结构等,DNA二级结构的形成就是一个大概率事件。形成的DNA二级结构会阻挡DNA Pol α、δ的移动,导致复制叉停顿。在酵母细胞的染色体DNA上,大约有2000-4000个能形成DNA二级结构的序列。在人细胞中,能形成DNA二级结构的数目大约有数百万个(~75万个G4结构,及数倍于G4的三股链结构等)。因此,DNA二级结构导致的复制叉停顿是一个经常发生的生物事件。在前导链或后随链的DNA模板上,如果有碱基损伤(每个人细胞每天有约105级别的这类损伤)或化学修饰,也会阻挡DNA Polα、δ、ε(DNA聚合酶的活性中心非常精巧,只能容纳AT或GC配对,但不能容纳AC或GT配对,以保证DNA复制的精准性),导致复制叉停顿。当DNA聚合酶被阻挡后,CMG解旋酶还要往前移动,这样就会导致CMG 解旋酶与DNA聚合酶物理上的分开,导致复制叉垮塌。
经过上千个实验室50年的不懈努力,checkpoint调控维持停顿DNA复制叉稳定的核心机制最终得到了阐明。即,1)停顿复制叉不稳定、倾向于垮塌的根本原因是:当DNA聚合酶被阻挡后,复制解旋酶与DNA聚合酶倾向于分开;2)checkpoint调控维持停顿复制叉稳定的核心机制是:降低复制解旋酶的活性,阻止复制解旋酶脱离复制叉或DNA聚合酶,维持两者之间在物理及生化功能上的紧密偶联,从而维持停顿复制叉稳定,并保持它的生物学功能(图2)。孔道春实验室于2019年发现的chromsfork调控,通过提高停顿复制叉周围的染色质结构紧密程度,也是为了阻挡复制解旋酶脱离复制叉,以维持复制叉稳定【Feng et al.(2019), PNAS, 116(29):14563- 14572】。
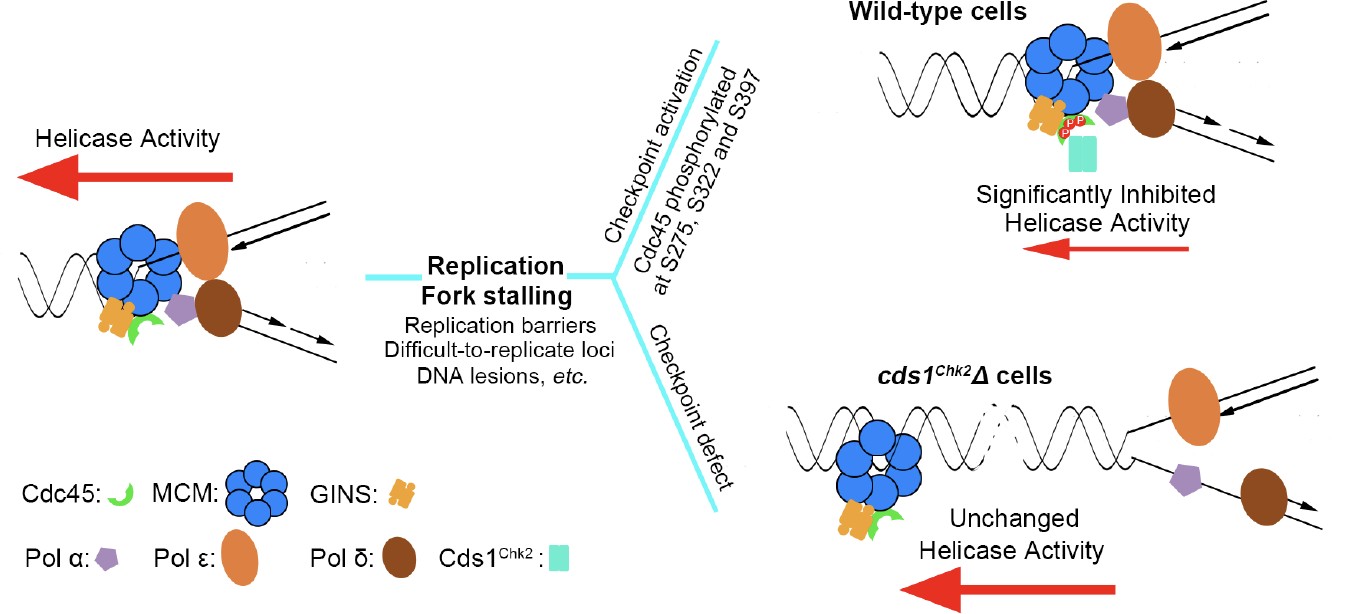
图2. Cds1Chk2 磷酸化复制解旋酶CMG 里的Cdc45 亚基,深度降低复制解旋酶活性,阻止复制解旋酶与DNA 聚合酶分开,从而稳定停顿复制叉
孔道春教授为该论文的通讯作者。刘阳博士和王露(2015级博士研究生)为该论文的共同第一作者。许鑫、袁越、张波、李泽阳、谢雨晨、闫睿、郑泽琦、北京大学生命科学学院纪建国教授、英国Sussex大学的Johanne M. Murray 和Antony M. Carr 教授也对本研究作出重要贡献。本工作得到了北大-清华生命科学联合中心、科技部国家重点研发计划、国家自然科学基金委、蛋白质与植物基因研究国家重点实验室,以及生命科学学院仪器中心(成像平台及流式平台)的大力支持。
