首先,为了系统性地研究斑马鱼尾部造血组织如何支持造血干细胞扩增、分化的机制,中国科学院动物研究所刘峰实验室利用三种转录组测序方法,特定类型群体细胞转录组、空间转录组6和单细胞转录组,详细解析了斑马鱼尾部造血组织的时空特性。不同发育时期特定细胞群体的转录组测序分析表明,造血干细胞与相应微环境细胞具有协同发育和共享基因表达的特点。空间转录组测序反映细胞位置信息,揭示了多种微环境因素对造血干细胞的不同调控方式,并实现了三维转录组重构斑马鱼造血器官。单细胞转录组数据更精细阐释尾部造血组织细胞类群、细胞特征及细胞间分子调控,并发现造血干祖细胞分化和细胞周期存在一定的关联性。多重转录组测序综合分析揭示了造血干细胞在尾部造血组织的扩增受到内源性和外源性信号的共同调控,鉴定了新的造血干细胞的标记基因,发现了影响造血干细胞扩增的信号通路,完善了造血干细胞-微环境细胞间分子调控网络。例如,染色质结构维持蛋白smchd1,通过调节造血干细胞增殖促进其扩增;微环境细胞来源的Integrin信号通路负向调控造血干细胞扩增。同时此项工作还构建了斑马鱼尾部造血组织多重转录组数据库http://www.picb.ac.cn/hanlab/ichtatlas/Home,可以检索任何基因在造血组织的表达谱,包括基因表达的时间、细胞类型及相对空间位置。
该工作不仅发现了造血干细胞特征基因、细胞状态与细胞分化的关系,阐释了微环境和造血干细胞互作的调控网络,更为血液系统研究提供了丰富的数据资源;同时,也将为造血干细胞的体外扩增和造血类器官的体外组装提供理论指导。该研究论文“A 3D atlas of hematopoietic stem and progenitor cell expansion by multi-dimensional RNA-seq analysis”于2019年4月30日在线发表于Cell Reports。
中国科学院动物研究所博士研究生薛媛媛和上海生科院计算生物学研究所博士研究生刘登辉为文章共同第一作者,中国科学院动物研究所刘峰研究员和上海生科院计算生物学研究所韩敬东研究员为文章共同通讯作者。中国科学院生物化学与细胞生物学研究所景乃禾研究员与中国科学院广州生物医药与健康研究院彭广敦研究员在空间转录组测序方面提供了大力支持。本研究得到了国家自然科学基金委、国家重点基础研究发展计划和中科院器官重建与制造战略性先导科技专项的资助。
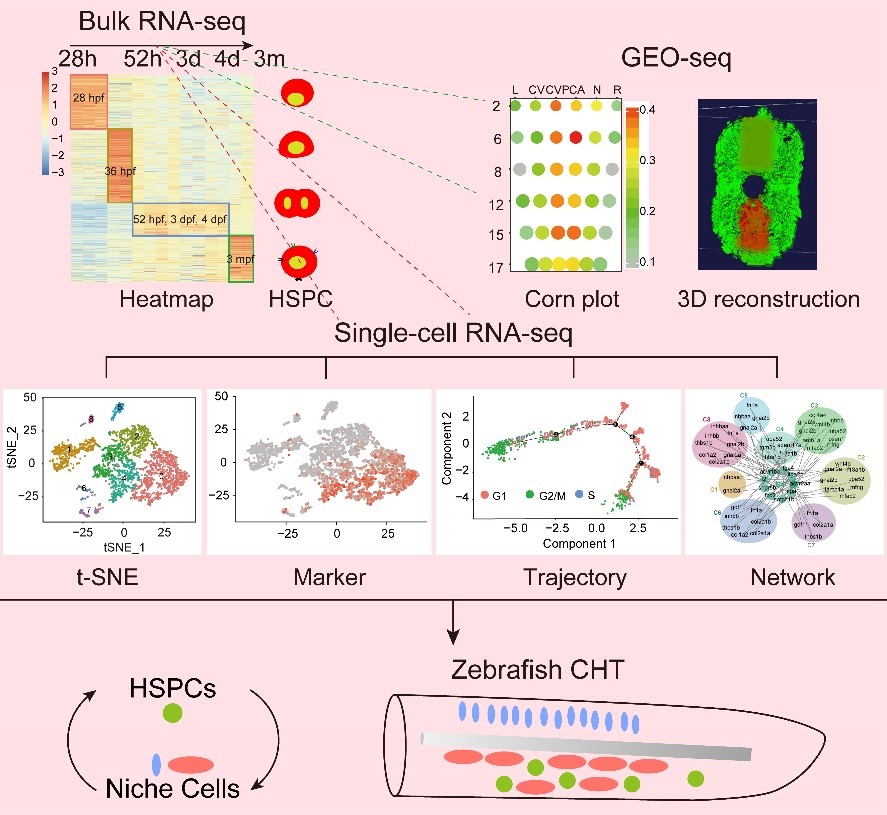
图1:多重转录组测序解析斑马鱼造血器官。多重转录组测序,包括:特定类型群体细胞转录组测序、空间转录组测序和单细胞转录组测序,揭示斑马鱼尾部造血组织的时空特性。
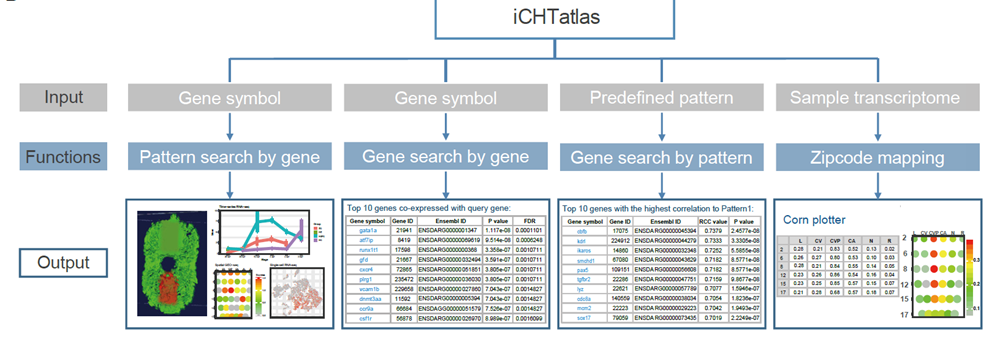
图2:斑马鱼造血器官转录组数据库
参考文献
1. Haas, S., Trumpp, A. & Milsom, M. D. Causes and Consequences of Hematopoietic Stem Cell Heterogeneity.Cell stem cell22, 627-638, doi:10.1016/j.stem.2018.04.003 (2018).
2. Zhang, Y. & Liu, F. Multidimensional Single-Cell Analyses in Organ Development and Maintenance.Trends in cell biology, doi:10.1016/j.tcb.2019.02.006 (2019).
3. Rowe, R. G., Mandelbaum, J., Zon, L. I. & Daley, G. Q. Engineering Hematopoietic Stem Cells: Lessons from Development.Cell stem cell18, 707-720, doi:10.1016/j.stem.2016.05.016 (2016).
4. Xue, Y.et al.The Vascular Niche Regulates Hematopoietic Stem and Progenitor Cell Lodgment and Expansion via klf6a-ccl25b.Developmental cell42, 349-362 e344, doi:10.1016/j.devcel.2017.07.012 (2017).
5. Tamplin, O. J.et al.Hematopoietic stem cell arrival triggers dynamic remodeling of the perivascular niche.Cell160, 241-252, doi:10.1016/j.cell.2014.12.032 (2015).
6. Chen, J.et al.Spatial transcriptomic analysis of cryosectioned tissue samples with Geo-seq.Nature protocols12, 566-580, doi:10.1038/nprot.2017.003 (2017).
| 附件下载: | |
