

中国科学院大学材料科学与光电技术学院, 北京 100049
2015年03月31日 收稿; 2015年05月08日 收修改稿
基金项目: 国家重点基础研究发展规划项目(2014CB931900)和国家自然科学基金(21274164, 21144001)资助
通信作者: E-mail:lixy@ucas.ac.cn
摘要: 通过分子动力学模拟,研究细胞色素c在氧化石墨烯表面的吸附行为.结果表明,细胞色素c和氧化石墨烯之间有较强的结合趋势.均方根涨落及回转半径分析表明,部分体系的吸附构象与原始构象比较接近,但P3系统有着较大的偏离.这些结果表明,在某些构象中,氧化石墨烯可能将细胞色素c结构破坏.
关键词: 细胞色素c氧化石墨烯吸附分子动力学
Adsorption of cytochrome c on graphene oxide: a molecular dynamic study
ZHANG Chi, HUANG Xuqi, LI Xiaoyi


College of Materials Science and Opto-electronics Technology, University of Chinese Academy of Sciences, Beijing 100049, China
Abstract: Through molecular dynamic simulations, we have investigated the adsorption behavior of cytochrome c on graphene oxide. Results indicate that graphene oxide has a great binding affinity towards cytochrome c. Analyses of root mean square deviation and gyration radius show that there is a large structural damage occurring in system P3 among all the systems. Our study shows that graphene oxide has the ability to deteriorate the native structure of cytochrome c.
Key words: cytochrome cgraphene oxideadsorptionmolecular dynamics
蛋白质在材料表面的吸附是一个基础问题,在解释材料细胞毒性领域有重要的意义.同时,它也是一个非常复杂的问题,因为它涉及蛋白质结构弹性、原子和分子间多种相互作用、水合化和去水合化等诸多因素.
细胞色素c(简称Cyt c)是一种广泛存在于线粒体内的球形蛋白.Cyt c在呼吸作用电子传输链中负责将电子通过氧化还原反应的形式,由bc1 complex运送到Cytochrome c oxidase.它是一种非常重要的蛋白质,因为它的功能影响到一系列重要生物化学过程,比如能量的产生与消耗、细胞分裂、细胞生长和凋亡等.Cyt c的一个特征是其亚铁血红素辅因子(heme)以共价键的形式连接蛋白质主干[1].由于其功能的重要性,以及广泛存在于各类细胞内这一事实,Cyt c吸引了大量研究者的关注.研究人员从电化学、电子转移和蛋白质折叠等多个角度[2-7]进行过深入的讨论.
氧化石墨烯是一种石墨氧化剥离后得到的单分子产物.由于具有极大的比表面积、一定的电子传输性能、易于化学修饰和良好的水溶性等性能,它被广泛应用于生物科技中,比如生物传感器[8]、细胞成像[9]和药物输运[8]等场合.
当纳米材料进入到细胞或血液时,机体的第一个反应就是蛋白质吸附[10].这种吸附可能会造成某些不利于细胞的作用[11-14].为了充分研究这个过程,研究人员使用了多种实验手段,比如荧光光谱[15]、中子散射[16]、圆二色谱[17]、傅里叶红外[18]、表面等离子体共振[19]、原子力显微镜[20]、X射线光谱[21]等.目前的研究已能充分解释纳米粒子对于蛋白质二级结构热力学等性质.尽管实验手段已经取得诸多进展,但是受限于其时间和空间的解析度,原子尺度的观察仍很缺乏.相比之下,分子动力学因其极精细的空间尺度和时间尺度,在纳米领域发挥重要作用.
本文选取Cyt c和氧化石墨烯作为吸附主体,采用分子动力学的研究手段.探讨了氧化石墨烯对Cyt c结构的影响,并解释了氧化石墨烯可能的生物毒性.
1 计算方法选用的马心细胞色素c晶体结构数据来自于Protein Data Bank,PDB ID 为1hrc[22].氧化石墨烯通过VMD软件建模得到[23],所采用的石墨烯模型来自Lerf等人的工作[24].氧化石墨烯片大小为70 ?×70 ?,碳氧比约为50.为了加速构象搜索,我们选取了常用的8个初始取向我们的氧化石墨烯模型垂直于z轴,因此采用Fe-S(Met80)键与z轴的夹角定义Cyt c的方向.P1构象中该角度设定为90°.依据P1构象,绕y轴旋转120°和240°得到P2和P4.依据P1构象绕x轴旋转,使Fe-S(Met80)键与z轴的夹角为28°、81°、155°、238°、315°,得到P6,P8,P7,P3,P5.更具体的初始取向选择说明参见我们之前的报道[25].所有初始构型中,Cyt c与氧化石墨烯之间的距离被设置为7 ?.氧化石墨烯的空间位置被固定.整个体系用尺寸为80 ?×80 ?×80 ?的周期性水盒子浸泡.水分子采用TIP3P[26]模型.为了模拟生物体系内的盐溶液浓度,并使体系保持电中性,我们加入了0.15 mol/L的钠离子和氯离子.
所有的模拟都使用NAMD 2.9[27]软件计算.采用NPT系综,温度为300 K,压力为1 atm,使用Berendsen方法控制恒温恒压[28].采用CHARMM27力场[29],对于heme部分采用Schulten等人报道的参数[30].氧化石墨烯碳原子采用CA类型原子,环氧基氧原子采用环氧乙烷氧原子参数,羟基氧原子采用乙醇羟基氧原子参数.排除策略为Scaled 1-4.使用SHAKE算法控制含氢原子共价键刚性.采用PME方法[31]处理长程静电相互作用,Grid Spacing设为1.0.范德华力截断值设置为12 ?.积分步长为2.0 fs.模拟时长为40ns.
2 结果与讨论2.1 吸附过程与最优吸附构象通过40 ns的模拟计算,所有的8个体系(P1、P2、P3、P4、P5、P6、P7、P8)蛋白都吸附在了氧化石墨烯的表面.这表明二者之间有较强的结合趋势.
Fig. 1
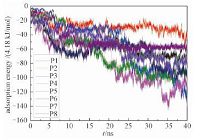 | Download: JPG larger image |
| 图 1 吸附能随时间的变化Fig. 1 Temporal evolution of adsorption energy | |
蛋白与氧化石墨烯之间有2种相互作用.一种是静电相互作用,一种是范德华相互作用.由于氧化石墨烯表面不带电荷,这里的全部非键相互作用等于范德华相互作用.对每个体系进行的吸附能计算,结果见图 1.结果表明,随着吸附的逐步进行,所有的体系结合能数值都在波动中下降,对应的结合越来越紧密.在最后2ns,所有体系都趋于稳定,表明吸附达到平衡.虽然从更长的时间尺度看,P3体系仍有下降趋势,但由于P3的吸附能已经是所有体系中最低的一个,可以判断其为最优吸附构象.从图 1中还可以看出,能量是阶梯式下降,台阶处意味着较大的能垒被克服,较大的构型变化正在发生.通过分析轨迹,发现这种构型变化,实质上是蛋白质残基的旋转与平移.
我们计算了体系最后2ns的平均吸附能,见表 1.从中可以看出,在体系P3中,吸附能绝对值最大,由此可以确定最优吸附构象存在于其中.
Table 1
| 表 1 吸附体系最后2 ns的平均回转半径Rg,吸附能,接触原子数Table 1 Average value of Rg,adsorption energy,and contact number | ||||||||||||||||||||||||||||||||||||||||||||
图 2是P3在40 ns时候的截图.包络heme基团的蛋白质二级结构已经被打开.Heme基团已经暴露出来.说明蛋白质经历了较大的形变,也就是说氧化石墨烯对Cyt c结构有着较强的影响.目前还没有实验报道类似的结果.我们的分子动力学数据可以用来预测.
Fig. 2
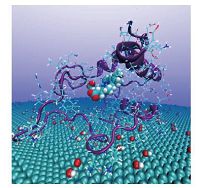 | Download: JPG larger image |
| 图 2 体系P3在40ns时的轨迹截图Fig. 2 Snapshot of system P3 at 40ns | |
除了相互作用能,还通过接触原子数,以及最小距离来衡量二者之间的吸附行为.我们定义接触原子数为在距离氧化石墨烯表面5 ?以内的非氢原子数.8个体系最终获得稳定的接触原子数和最小距离.体系在最后2ns的平均接触原子数列在表 1中.P3的接触原子数最多,而且其最短距离最小.由动力学轨迹我们发现,原子吸附在表面后,在观察时长范围内都没有脱离.这说明二者之间相互作用较强,吸附不可逆.
我们定义Cyt c和氧化石墨烯之间的平均接触表面积(ACSA)为ACSA= <1/2[(SASAProtein+SASAGraphene oxide)-SASAComplex>last 2ns.其中,SASA表示溶液可及表面积.随着系统动力学过程的进行,所有体系的ACSA都在逐渐增大,并最终趋于稳定.将ACSA与吸附能做图,可以发现二者之间呈线性关系.这直接表明二者之间的正比例关系,也就是说,接触面积越大,吸附作用越强.
Fig. 3
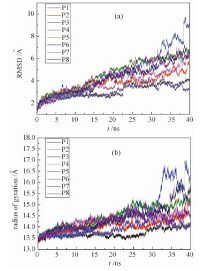 | Download: JPG larger image |
| 图 3 (a)均方根涨落RMSD随时间的变化;(b)均方回转半径Rg随时间的变化Fig. 3 Temporal evolution of RMSD (a) and Rg (b) | |
2.2 吸附对于蛋白质结构的影响蛋白质的生物功能是由其残基序列以及多级结构产生的.如果结构受到破坏,那么功能也会丧失.通过计算均方根涨落RMSD以及回转半径Rg我们分析了氧化石墨烯对Cyt c的结构的影响.RMSD如图 3(a)所示,P3体系RMSD较大,峰值达到10?附近,说明Cyt c的结构受到的较大程度的影响,与轨迹观察相一致.观察其动力学轨迹我们发现,由3个α螺旋包裹的heme crevice已经被拉开,将heme基团暴露在溶剂中.由于heme是Cyt c蛋白的核心活性位点,这种结构的暴露导致Cyt c蛋白功能的丧失.其他几个体系的RMSD均为6?左右,属于较小的波动.Rg如图 3(b)所示,与RMSD类似,P3的值最大,达到17?.其他体系低于15.5?.Cyt c本身在纯水溶液中的Rg为13.3?,因此可以认为除P3外,其他体系的吸附构象与原始构象都比较接近.这些结果表明,在某些构象中,氧化石墨烯可能将Cyt c结构破坏.也就是说,氧化石墨烯有着潜在的蛋白生物毒性.
3 结论本文采用分子动力学方法,研究了细胞色素c在氧化石墨烯表面的吸附行为.通过40 ns的模拟计算,在所有体系中蛋白都吸附在了氧化石墨烯的表面.这表明二者之间有着较强的结合趋势.观察轨迹发现,随着系统动力学过程的进行,所有体系的接触表面积都在逐渐增大,并最终趋于稳定.将接触面积与吸附能做图,可以发现二者之间呈线性关系.均方根涨落及回转半径分析表明,部分体系的吸附构象与原始构象都比较接近,但P3系统有着较大的偏离.这些结果表明,在某些构象中,氧化石墨烯可能将Cyt c结构破坏.
本文的计算结果是在中国科学院计算机网络信息中心超级计算中心的“元”超级计算机上得到的.
参考文献
| [1] | Stevens J M. Cytochrome c as an experimental model protein[J].Metallomics, 2011, 3(4):319–322.DOI:10.1039/c0mt00089b |
| [2] | Weinkam P, Zimmermann J, Romesberg F E, et al. The folding energy landscape and free energy excitations of cytochrome c[J].Accounts of Chemical Research, 2010, 43(5):652–660.DOI:10.1021/ar9002703 |
| [3] | Winkler J R. Cytochrome c folding dynamics[J].Current Opinion in Chemical Biology, 2004, 8(2):169–174.DOI:10.1016/j.cbpa.2004.02.009 |
| [4] | Stevens J M, Uchida T, Daltrop O, et al. Covalent cofactor attachment to proteins: cytochrome c biogenesis[J].Biochemical Society Transactions, 2005, 33(Pt 4):792–795. |
| [5] | Allen J W, Daltrop O, Stevens JM, et al. C-type cytochromes: diverse structures and biogenesis systems pose evolutionary problems[J].Philos Trans R Soc Lond B Biol Sci, 2003, 358(1429):255–266.DOI:10.1098/rstb.2002.1192 |
| [6] | Diekert K, de Kroon A I, Ahting U, et al. Apocytochrome c requires the TOM complex for translocation across the mitochondrial outer membrane[J].EMBO Journal, 2001, 20(20):5626–5635.DOI:10.1093/emboj/20.20.5626 |
| [7] | Bashir Q, Volkov A N, Ullmann G M, et al. Visualization of the encounter ensemble of the transient electron transfer complex of cytochrome c and cytochrome c peroxidase[J].Journal of the American Chemical Society, 2010, 132(1):241–247.DOI:10.1021/ja9064574 |
| [8] | Feng L, Chen Y, Ren J, et al. A graphene functionalized electrochemical aptasensor for selective label-free detection of cancer cells[J].Biomaterials, 2011, 32(11):2930–2937.DOI:10.1016/j.biomaterials.2011.01.002 |
| [9] | Peng C, Hu W, Zhou Y, et al. Intracellular imaging with a graphene-based fluorescent probe[J].Small, 2010, 6(15):1686–1692.DOI:10.1002/smll.v6:15 |
| [10] | Wei T, Carignano M A, Szleifer I. Lysozyme adsorption on polyethylene surfaces: why are long simulations needed?[J].Langmuir, 2011, 27(19):12074–12081.DOI:10.1021/la202622s |
| [11] | Heinz H, Farmer B L, Pandey R B, et al. Nature of molecular interactions of peptides with gold, palladium, and Pd-Au bimetal surfaces in aqueous solution[J].Journal of the American Chemical Society, 2009, 131(28):9704–9714.DOI:10.1021/ja900531f |
| [12] | Yang W, Xue H, Li W, et al. Pursuing "zero" protein adsorption of poly(carboxybetaine) from undiluted blood serum and plasma[J].Langmuir, 2009, 25(19):11911–11916.DOI:10.1021/la9015788 |
| [13] | Dringen R, Koehler Y, Derr L, et al. Adsorption and reduction of glutathione disulfide on alpha-Al2O3 nanoparticles: experiments and modeling[J].Langmuir, 2011, 27(15):9449–9457.DOI:10.1021/la201856p |
| [14] | Nel AE, Madler L, Velegol D, et al. Understanding biophysicochemical interactions at the nano-bio interface[J].Nature Materials, 2009, 8(7):543–557.DOI:10.1038/nmat2442 |
| [15] | Benesch J, Hungerford G, Suhling K, et al. Fluorescence probe techniques to monitor protein adsorption-induced conformation changes on biodegradable polymers[J].Journal of Colloid and Interface Science, 2007, 312(2):193–200.DOI:10.1016/j.jcis.2007.03.016 |
| [16] | Lu JR, Su TJ, Thirtle PN, et al. The denaturation of lysozyme layers adsorbed at the hydrophobic solid/Liquid surface studied by neutron reflection[J].Journal of Colloid and Interface Science, 1998, 206(1):212–223.DOI:10.1006/jcis.1998.5680 |
| [17] | Billsten P, Wahlgren M, Arnebrant T, et al. Structural changes of T4 lysozyme upon adsorption to silica nanoparticles measured by circular dichroism[J].Journal of Colloid and Interface Science, 1995, 175(1):77–82.DOI:10.1006/jcis.1995.1431 |
| [18] | Wei T, Kaewtathip S, Shing K. Buffer effect on protein adsorption at liquid solid interface[J].The Journal of Physical Chemistry C, 2009, 113(6):2053–2062.DOI:10.1021/jp806586n |
| [19] | Li G, Xue H, Gao C, et al. Nonfouling Polyampholytes from an lon-pair comonomer with biomimetic adhesive groups[J].Macromolecules, 2010, 43(1):14–16.DOI:10.1021/ma902029s |
| [20] | Kim D T, Blanch H W, Radke C J. Direct imaging of lysozyme adsorption onto mica by atomic force microscopy[J].Langmuir, 2002, 18(15):5841–5850.DOI:10.1021/la0256331 |
| [21] | Zhong J, Song L, Meng J, et al. Bio-nano interaction of proteins adsorbed on single-walled carbon nanotubes[J].Carbon, 2009, 47(4):967–973.DOI:10.1016/j.carbon.2008.11.051 |
| [22] | Bushnell G W, Louie G V, Brayer G D. High-resolution three-dimensional structure of horse heart cytochrome c[J].Journal of Molecular Biology, 1990, 214(2):585–595.DOI:10.1016/0022-2836(90)90200-6 |
| [23] | Humphrey W, Dalke A, Schulten K. VMD: visual molecular dynamics[J].Journal of Molecular Graphics, 1996, 14(1):33–38.DOI:10.1016/0263-7855(96)00018-5 |
| [24] | Lerf A, He H, Forster M, et al. Structure of graphite oxide revisited[J].The Journal of Physical Chemistry B, 1998, 102(23):4477–4482.DOI:10.1021/jp9731821 |
| [25] | Hu B, Ge Z, Li X. Interface adsorption taking the most advantageous conformation for electron transfer between graphene and cytochrome c[J].Journal of Nanoscience and Nanotechnology, 2015, 15(7):4863–4869.DOI:10.1166/jnn.2015.9853 |
| [26] | Jorgensen W L, Chandrasekhar J, Madura J D, et al. Comparison of simple potential functions for simulating liquid water[J].Journal of Chemical Physics, 1983, 79(2):926–935.DOI:10.1063/1.445869 |
| [27] | Phillips J C, Braun R, Wang W, et al. Scalable molecular dynamics with NAMD[J].J Comput Chem, 2005, 26(16):1781–1802.DOI:10.1002/(ISSN)1096-987X |
| [28] | Berendsen H J C, Postma J P M, van Gunsteren WF, et al. Molecular dynamics with coupling to an external bath[J].Journal of Chemical Physics, 1984, 81(8):3684–3690.DOI:10.1063/1.448118 |
| [29] | MacKerell A D, Bashford D, Bellott M, et al. All-atom empirical potential for molecular modeling and dynamics studies of proteins[J].J Phys Chem B, 1998, 102(18):3586–3616.DOI:10.1021/jp973084f |
| [30] | Autenrieth F, Tajkhorshid E, Baudry J, et al. Classical force field parameters for the heme prosthetic group of cytochrome c[J].J Comput Chem, 2004, 25(13):1613–1622.DOI:10.1002/jcc.v25:13 |
| [31] | Darden T, York D, Pedersen L. Particle mesh ewald: an NlogN method for ewald sums in large systems[J].Journal of Chemical Physics, 1993, 98(12):10089–10092.DOI:10.1063/1.464397 |
