

中国科学院大学材料科学与光电技术学院, 北京 100049
2017年04月10日 收稿; 2017年05月19日 收修改稿
基金项目: 国家自然科学基金(21274164)和973项目(2014CB931900)资助
通信作者: 李晓毅,E-mail:lixy@ucas.ac.cn
摘要: 通过分子动力学模拟,研究DNA/B-PDEAEA纳米颗粒的自组装过程。模拟结果表明,在自组装过程中静电相互作用是主要驱动力;在相同氮磷比的条件下,随着DNA碱基对数目的增加,更易于形成稳定的纳米颗粒。在相同碱基对数目的条件下,随着氮磷比的增加,更趋向于形成稳定的纳米颗粒。
关键词: DNA自组装分子动力学模拟
Molecular dynamics simulation of self-assembly process of DNA and B-PDEAEA
HUANG Xuqi, LI Xiaoyi


College of Materials Science and Opto-electronics Technology, University of Chinese Academy of Sciences, Beijing 100049, China
Abstract: The self-assemble processes of DNA/B-PDEAEA nanoparticles are investigated by means of molecular dynamics simulations. The simulation results show that the electrostatic interaction force is the main driving force for the self-assembly process. At the same N/P ratio, stable nanoparticles form easier when the number of DNA base pairs is larger. With the same number of base pairs, stable nanoparticles form easier at larger N/P ratio.
Key words: DNAself-assemblymolecular dynamics simulation
自组装[1-8]是在纳米尺度或微观尺度上构建有序结构的一个自发过程,在自组装过程中,小分子主要借助非共价键相互作用形成纳米颗粒。随着科技的发展,纳米生物技术[9-12]备受关注,其中纳米载药系统是国际生物技术领域的前沿课题,在治疗一些疾病如癌症、寄生虫感染等方面,有着广泛的应用和产业化的前景。研究载体材料与核酸药物的复合过程、络合强度、纳米复合物的稳定性及对核酸药物的保护作用有着重要的意义。针对常规季胺化聚合物包裹DNA进入细胞后难以解离导致转染效率低的问题,给予肿瘤细胞内高浓度的活性氧自由基(reactive oxygen species, ROS),Liu等[13]设计了4-溴甲基苯硼酸季胺化的阳离子聚合物(B-PDEAEA)载体。这种载体不仅可以稳定地结合DNA,而且在高活性氧化自由基环境下失去正电荷有利于释放出游离的DNA。这种载体在细胞水平相比于PEI的转染效率提高1~2个数量级,具有较高的抗血清能力,且毒性较小。因此,设计所得药物的抗癌效率远高于传统的抗癌药物。为了研究自组装过程,研究人员用许多先进实验方法来分析结构变化,如X射线衍射(XRD)[14]、核磁共振波谱(NMR)[15]、扫描隧道显微镜(STM)[16]、激光散射[17]等。尽管DNA与B-PDEAEA的纳米粒子体系在实验上已经取得了一些成果,但在分子水平上对其形成机理的研究还很少。计算机模拟为研究复杂体系的自组装行为提供了另一种途径,通过模拟可以研究DNA与B-PDEAEA的自组装过程。
1 计算方法4-溴甲基苯硼酸季胺化的阳离子聚合物的单体结构如图 1所示,4-溴甲基苯硼酸季胺化的阳离子聚合物的单体模型通过Gaussian09[18]构建并进行结构优化。通过Gaussian09计算其RESP电荷,硼酸的力场参数来源于Tafi等[19]和Rohit等[20]的工作;DNA通过VMD软件[21]建模得到。
Fig. 1
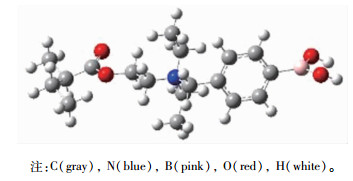 | Download: JPG larger image |
| 图 1 B-PDEAEA单体结构 Fig. 1 Monomer structure of B-PDEAEA 图 1 B-PDEAEA单体结构 Fig. 1 Monomer structure of B-PDEAEA --> | |
所有的模拟都使用NAMD2.9软件[22]计算。本文采用NVT系统,温度为300 K,压力为1 atm。采用CHARMM36力场,应用隐式水溶剂,排除策略为Scale 1-4,使用SHAKE算法控制含氢原子的共价键为刚性。范德华力截断值设置为16 ?,积分步长为2.0 fs,模拟时间为10 ns。采用隐式水溶剂,可以减少体系的原子数,忽略溶剂的摩擦效应,加快构象的变化。为了研究DNA的碱基对数目和体系的N/P对自组装的影响,选取6个体系进行研究,如表 1所示。
Table 1
| 表 1 模拟系统Table 1 Simulation system | ||||||||||||||||||||||||||||||||
2 结果与讨论通过10 ns的模拟计算,6个体系中P3、P5、P6自组装形成稳定聚集体,而P1、P2、P4没有形成稳定的聚集体。从分子结构分析,4-溴甲基苯硼酸季胺化的阳离子聚合物中季胺基团携带正电荷,而DNA携带有少量的阴离子。因此,静电相互作用在自组装过程中占有举足轻重的作用。图 2给出体系中各部分的非键相互作用。
Fig. 2
 | Download: JPG larger image |
| 图 2 VDW和ELE所占比例 Fig. 2 Proportions of VDW and ELE 图 2 VDW和ELE所占比例 Fig. 2 Proportions of VDW and ELE --> | |
由图 2中可知静电相互作用(electrostatic interaction,ELE)所占比重超过99%,静电相互作用是该自组装过程的主要驱动力,而范德华相互作用(Van der Waals,VDW)的贡献非常小,只占0.31%。在相同的碱基对的情况下,由于季胺基团携带正电荷,随着N/P的增加,静电的相互作用力显著增强,体系越容易趋向于形成稳定的聚集体。当N/P相同时,随着碱基对数目的增加,DNA携带的负电荷数目少量增加,静电相互作用增强。
实验结果[13]表明,在一定范围内,当碱基对数目较少时,不易于形成稳定的聚集体,随着N/P和碱基对数目的增加,体系趋向于形成稳定的聚集体。模拟体系的最初构型中,DNA分子在体系的中心,B-PDEAEA分子随机地分散在体系中,随着模拟时间的延长,不同的体系呈现不同的状态,如图 3所示。
Fig. 3
 | Download: JPG larger image |
| 图 3 体系在10 ns时的截图 Fig. 3 Snapshots of system at 10 ns 图 3 体系在10 ns时的截图 Fig. 3 Snapshots of system at 10 ns --> | |
P1、P2、P4没有形成稳定的聚集体,仍然处于较为松散的状态,其中P1的形态最为松散;P3、P5、P6都能够自组装形成聚集体。通过对比6个体系,当碱基对数目不变时,随着N/P的增加,体系越容易趋向于形成稳定的聚集体; 当碱基对数目不变时,随着N/P的增加,体系也更趋向于形成稳定的聚集体。表明B-PDEAEA分子随着DNA的碱基对数目和N/P的增加,在水溶液中更容易发生聚集。随后,聚集体构型逐步调整达到平衡,形成稳定的圆形纳米球。
为了分析自组装体系的聚集状态,本文分析均方回转半径与模拟时间的变化曲线。如图 4(a)所示,P1、P2、P4体系中B-PDEAEA分子的Rg值呈现波动上升状态, 其中P1波动幅度最大;P3、P5、P6体系中B-PDEAEA分子回转半径的变化先逐渐降低后处于上下震动状态,没有大幅度的变化,说明P3、P5、P6体系逐渐趋向于稳定。如图 4(b)所示,DNA回转半径基本没有变化,Rg值一直稳定在某个值左右,表明不论模拟时间怎么变化,DNA的结构基本不会受到影响。
Fig. 4
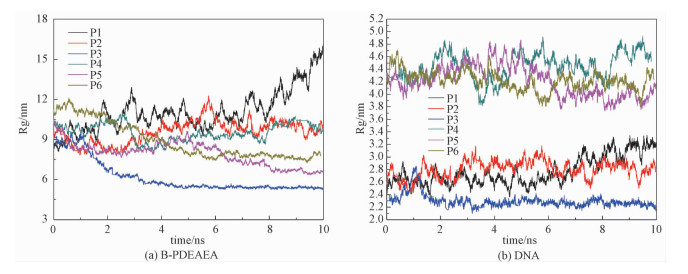 | Download: JPG larger image |
| 图 4 均方回转半径随时间的变化 Fig. 4 Temporal evolution of Rg 图 4 均方回转半径随时间的变化 Fig. 4 Temporal evolution of Rg --> | |
为了更好地研究自组装聚集体中B-PDEAEA的分布状态,分析B-PDEAEA分子围绕DNA的质心位置的径向分布函数,从图 5可以发现与上述相似的结果。P1、P2、P4的曲线有多个峰值,B-PDEAEA分子分布较为分散,其中P1的径向分布函数有多个峰值,而且最大峰值对应的距离值最大,因此体系的分散程度最大;P3、P5、P6只有一个峰值,峰值对应的距离值较小,B-PDEAEA分子环绕在DNA附近,聚集度较高。在相同的碱基对数目情况下,随着N/P的增加,体系更趋向于形成稳定的聚集体;在相同的N/P的情况下,随着碱基对数目的增加,P4、P5体系中的聚集程度相对P1、P2有一定的提升。
Fig. 5
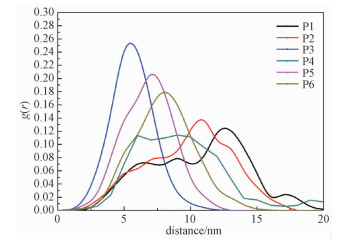 | Download: JPG larger image |
| 图 5 B-PDEAEA分子围绕DNA的质心位置的径向分布函数 Fig. 5 Radial distribution function of B-PDEAEA from the center of mass of DNA 图 5 B-PDEAEA分子围绕DNA的质心位置的径向分布函数 Fig. 5 Radial distribution function of B-PDEAEA from the center of mass of DNA --> | |
为进一步说明不同体系中B-PDEAEA的分布情况,分析最后1 ns时间内6个体系质心距离随时间的变化。如图 6所示,P1、P4体系的质心距离波动幅度较大,其他体系没有出现大幅波动的情况;P3的质心距离值最小,最为稳定。
Fig. 6
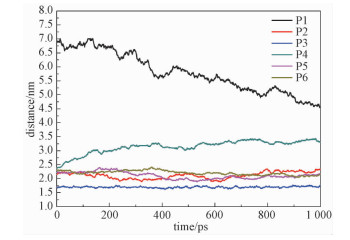 | Download: JPG larger image |
| 图 6 B-PDEAEA和DNA质心距离随时间的变化 Fig. 6 Temporal evolution of centroid distances for B-PDEAEA and DNA 图 6 B-PDEAEA和DNA质心距离随时间的变化 Fig. 6 Temporal evolution of centroid distances for B-PDEAEA and DNA --> | |
3 结论本文采用分子动力学的方法,研究B-PDEAEA与DNA的自组装过程。通过10 ns的模拟计算可知,P1、P2、P4没有形成稳定的聚集体,仍然处于较为松散的状态;P3、P5、P6都能够自组装形成聚集体,该结果与已报道过的实验结果[13]完全相符。通过两组对照实验表明,在相同DNA碱基对数目的情况下,随着N/P增加,体系更趋向于形成聚集体。在一定范围内,相同的N/P的情况下,随着DNA碱基对数目增加,体系更趋向于形成自组装纳米粒子。因此,N/P和DNA碱基对数目是影响体系自组装的重要因素。
本文的计算结果是在中国科学院计算机网络信息中心超级计算中心的“元”超级计算机上得到的。
参考文献
| [1] | Whitesides G M, Grzybowski B. Self-assembly at all scales[J].Science, 2002, 295:2418–2421.DOI:10.1126/science.1070821 |
| [2] | Whrtesides G M, Mathias J P, Seto C T. Molecular self-assembly and nanochemistry:a chemical strategy for the synthesis of nanostructures[J].Science, 1991, 254:1312–1319.DOI:10.1126/science.1962191 |
| [3] | Mackenzie R, Booth J, Alexander C, et al. Multiscale modeling of drug-polymer nanoparticle assembly identifies parameters influencing drug encapsulation efficiency[J].Journal of Chemical Theory and Computation, 2015, 11(6):2705–2713.DOI:10.1021/ct501152a |
| [4] | Kraft J F, Vestergaard M, Schiott B, et al. Modeling the self-assembly and stability of DHPC micelles using atomic resolution and coarse grained MD simulations[J].Journal of Chemical Theory and Computation, 2012, 8(5):1556–1569.DOI:10.1021/ct200921u |
| [5] | Nguyen T D, Zhang Z, Glotzer S C. Molecular simulation study of self-assembly of tethered Ⅴ-shaped nanoparticles[J].The Journal of Chemical Physics, 2008, 129(24):1–10. |
| [6] | Li S D, Chen Y C, Hackett M J, et al. Tumor-targeted delivery of siRNA by self-assembled nanoparticles[J].Molecular Therapy, 2008, 16(1):163–169.DOI:10.1038/sj.mt.6300323 |
| [7] | Sharma S, Juffer A H. An atomistic model for assembly of transmembrane domain of T cell receptor complex[J].Journal of the American Chemical Society, 2013, 135(6):2188–2197.DOI:10.1021/ja308413e |
| [8] | Whitelam S, Bon S A. Self-assembly of amphiphilic peanut-shaped nanoparticles[J].The Journal of Chemical Physics, 2010, 132(7):074901.DOI:10.1063/1.3316794 |
| [9] | Wang A Z, Langer R, Farokhzad O C. Nanoparticle delivery of cancer drugs[J].Annual Review of Medicine, 2012, 63:185–198.DOI:10.1146/annurev-med-040210-162544 |
| [10] | Puri S, Kallinteri P, Higgins S, et al. Drug incorporation and release of water soluble drugs from novel functionalized poly(glycerol adipate) nanoparticles[J].Journal of Controlled Release, 2008, 125(1):59–67.DOI:10.1016/j.jconrel.2007.09.009 |
| [11] | Jelonek K, Kasperczyk J, Li S, et al. Controlled poly(l-lactide-co-trimethylene carbonate) delivery system of cyclosporine A and rapamycine:the effect of copolymer chain microstructure on drug release rate[J].International Journal of Pharmaceutics, 2011, 414(1/2):203–209. |
| [12] | Shi Y C, Li X Y. Biomedical applications and adverse health effects of nanomaterials[J].Nanoscience and Nanotechnology, 2012, 12(11):8231–8240.DOI:10.1166/jnn.2012.6631 |
| [13] | Liu X, Xiang J, Zhu D, et al. Fusogenic reactive oxygen species triggered charge-reversal vector for effective gene delivery[J].Advanced Materials, 2016, 28(9):1743–1752.DOI:10.1002/adma.201504288 |
| [14] | Brown S P, Schnell I, Brand J D, et al. An investigation of π-π packing in a columnar hexabenzocoronene by fast magic-angle spinning and double-quantum 1H solid-state NMR spectroscopy[J].Journal of American Chemical Society, 1999, 121:6712–6718.DOI:10.1021/ja990637m |
| [15] | Mcmahon C A, Hawrylak B, Gerrard M D, et al. Calorimetric and NMR investigations of the micellar properties of sodium dodecyl sulfate in aqueous mixtures of isomeric butanediols[J].Langmuir, 1999, 15(2):429–436.DOI:10.1021/la971093+ |
| [16] | Markey L, Stievenard D, Devos A, et al. STM observations of self-assembled ID and 2D nanoclusters of aromatic cryptand molecules deposited on highly oriented pyrolytic graphite[J].Supramolecular Science, 1997, 4:3–4.DOI:10.1016/S0968-5677(96)00045-4 |
| [17] | Huang Y X, Tan R C, Li Y L, et al. Effect of salts on the formation of C8-lecithin micelles in aqueous solution[J].Journal of Colloid and Interface Science, 2001, 236(1):28–34.DOI:10.1006/jcis.2000.7278 |
| [18] | Frich M J, Truck G W, Schelegel H B, et al. Gaussion09[CP]. Gaussian Inc, Wallingford CT, 2009. |
| [19] | Tafi A, Agamennone M, Tortorella P, et al. AMBER force field implementation of the boronate function to simulate the inhibition of beta-lactamases by alkyl and aryl boronic acids[J].European Journal of Medicinal Chemistry, 2005, 40(11):1134–1142.DOI:10.1016/j.ejmech.2005.06.011 |
| [20] | Rohit T K M, Pavlovicz R, Li C L, et al. Carborane clusters in computational drug design:a comparative docking evaluation using AutoDock, FlexX, Glide, and Surflex[J].Journal of Chemmical Information and Modeling, 2009, 49:1581–1589.DOI:10.1021/ci900031y |
| [21] | Humphrey W, Dalke A, Schulten K. VMD-visual molecular dynamic[J].J Molec Graphics, 1996, 14:33–38.DOI:10.1016/0263-7855(96)00018-5 |
| [22] | Phillips J C, Braun R, Wang W, et al. Scalable molecular dynamic with NAMD[J].J Comput Chem, 2005, 26(16):1781–1802.DOI:10.1002/(ISSN)1096-987X |
