转基因生物(GMO)的分子特征主要包括外源DNA在受体基因组上的整合位点、完整插入序列和结构、旁侧序列和拷贝数等信息。分子特征解析是转基因生物安全评价的核心内容之一,每个转基因生物品系必须提供准确、完善的分子特征数据才可能被批准商业化生产应用。因此,准确识别和解析转基因生物的分子特征对于我国转基因生物的商业化应用至关重要。但是,现有的转基因技术都是通过随机整合方式将外源DNA转入受体物种的基因组上的,整合方式及机制尚不清晰,导致外源DNA整合的分子特征识别和解析非常困难。特别是,对具有复杂重排、倒位和串联重复等复杂结构的大DNA片段整合的分析技术方法十分缺乏。为此,上海交通大学国家转基因生物分子特征验证测试中心开发了一种通用型的适合于多种转基因生物的分子特征破译策略LIFE-Seq。
该研究首先从团队前期建立的转基因生物检测方法数据库(GMDD)中,筛选主要常用的转基因元件、外源基因、物种内标准基因等构建序列数据集,设计了一套高覆盖率的寡核苷酸探针库,可以覆盖99.1%的商业化转基因作物;然后利用液相杂交富集捕获转基因生物基因组中含有外源DNA序列的大片段;最后利用PacBio测序获得捕获的大片段DNA的具体序列信息,构建了长片段序列分析算法,实现对外源DNA整合位点、方式、结构和旁侧序列的完整破译。
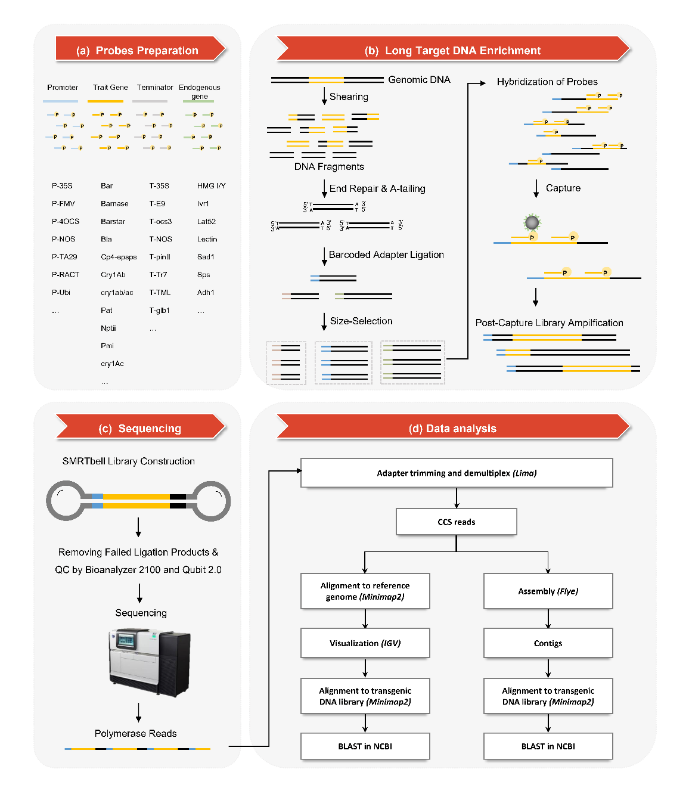
图1.LIFE-Seq 工作原理示意图
实验以6种转基因玉米、大豆、水稻和油菜商业化应用的转化体为实验材料,设计了不同转基因含量的测试样品以及非转基因对照样品,系统测试了LIFE-Seq方法的可行性和准确性。每个测试样本生成约 26,352 个高保真 CCS reads,平均长度约 6275 bp,所获得的CCS reads成功覆盖了相应的植物内源参考基因,说明系统可以对目标DNA片段有效富集。同时覆盖植物参考基因组序列和转基因外源DNA文库的CCS reads被筛选出作为候选CCS reads进行完整的插入结构解析。解析结果表明利用LIFE-Seq 成功破译了5个转基因转化体(NK603玉米、MON810玉米、TT51-1水稻、GTS40-3-2 大豆和 RT73油菜)的外源DNA整合等分子特征。在RF2转化体含量只有1.0%的混合样本中,也分析获得了RF2转化体的部分整合位点分子特征数。例如,在NK603玉米样本中,直接获得了含有完整的7462bp外源DNA整合结构和插入位点旁侧序列的CCS read,序列信息直接阐明了外源DNA插入到受体玉米基因组的Chr06 61665252处,有3bp的缺失。MON810玉米的结果显示直接获得了CCS reads含有完整的外源DNA全部整合结构、插入位点旁侧序列和受体基因组在整合位点附近的重排结构。转基因NK603玉米和MON810玉米外源DNA的整个插入结构见图2所示。
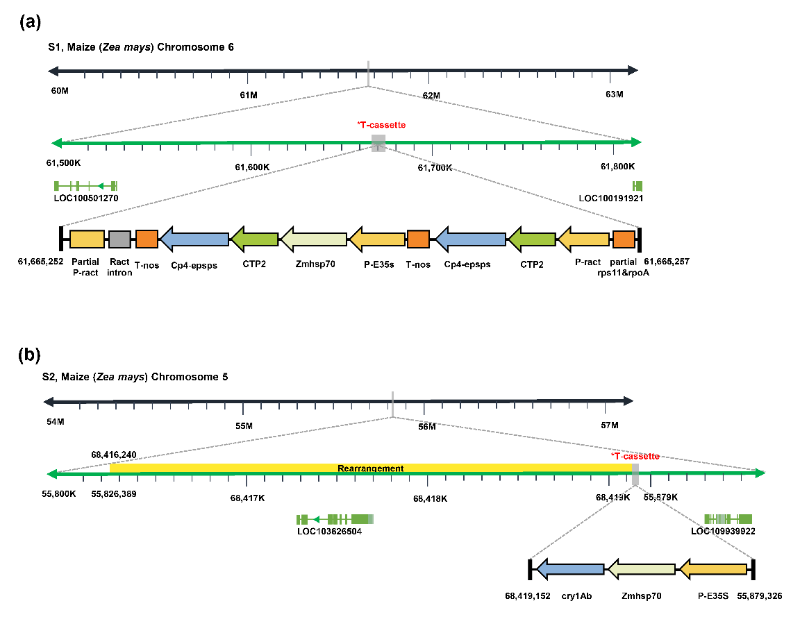
图2.转基因玉米NK603(S1)和MON810(S2)中转基因整合结构示意图
研究结果表明,LIFE-Seq成功对多种转基因作物转化体的外源DNA整合分子特征进行了清晰、准确的解析。与Southern blot、Sanger测序、重测序等技术相比,外源DNA整合数据解析的完整性和准确性高,序列分析接单、通用性强、成本低,特别适用于含有复杂外源DNA整合结构的转基因生物的分子特征解析。同时,LIFE-Seq也可以进一步用于未知转基因产品的检测和分子特征解析、及复杂基因组DNA结构的分析。
杨立桃课题组近年来在转基因生物分子特征识别和检测方面取得了系列进展(J Agric Food Chem, 2021, 69, 1705?1713; ACS Agric. Sci. Technol. 2021, 1, 390?399; Food Chemistry: Molecular Sciences, 2022. 4 :100061),重点开发了系列转基因作物外源DNA插入整合高通量、通用型精准分析新方法和新技术。该研究获得了国家转基因生物新品种培育重大专项和上海高校****(****)项目经费的支持。
论文链接:https://onlinelibrary.wiley.com/doi/10.1111/pbi.13776
