

1. 东北大学 医学与生物信息工程学院,辽宁 沈阳 110169;
2. 中国医科大学附属盛京医院,辽宁 沈阳 110801;
3. 深圳技术大学 健康与环境工程学院,广东 深圳 518118
收稿日期:2022-06-28
基金项目:国家自然科学基金资助项目(62071311);国家重点研发计划项目(2018YFC1002900)。
作者简介:杨超然(1988-),男,辽宁沈阳人,东北大学博士研究生;
康雁(1964-),男,辽宁沈阳人,东北大学教授,博士生导师。
摘要:在产前超声筛查过程中,为了能够帮助医生在丘脑标准平面上快速、精确地测量胎儿头围,提出一种新颖的双分支卷积神经网络直接分割胎儿颅骨边界,2个分支通过共享层相互促进,有效地提高了颅骨边界的分割精度,特别对局部不清晰或者不连续的边界仍然有着较好的分割效果,具有较高的鲁棒性.本方法的测量过程不需要过多的后处理操作,并且模型属于轻量级网络,便于部署.该方法应用在Grand-Challenge中的HC18数据集及从医院采集的300例数据上,均取得了较好的结果,对比其他主流分割网络如U-Net,Res-U-Net,U-Net++,CE-Net等,所提方法具有更高的分割精度及更小的测量误差.
关键词:产前超声头围测量边界分割卷积神经网络轻量级
Automatic Measurement Method for Fetal Head Circumference Based on Convolution Neural Network
YANG Chao-ran1, LIAO Shan-shan2, CHEN Da1, KANG Yan1,3


1. School of Medicine and Biological Information Engineering, Northeastern University, Shenyang 110169, China;
2. Shengjing Hospital of China Medical University, Shenyang 110801, China;
3. School of Health Science Environmental Engineering, Shenzhen Technology University, Shenzhen 518118, China
Corresponding author: KANG Yan, E-mail: kangyan@sztu.edu.cn.
Abstract: In prenatal ultrasound screening, in order to help doctors measure the fetal head circumference quickly and accurately on the standard plane of the thalamus, a novel two-branch convolution neural network is proposed to directly segment the fetal skull boundary. The two branches promote each other through the shared layer, which can improve the segmentation accuracy of the skull boundary effectively. In particular, the proposed method has good segmentation effects and high robustness for locally unclear or discontinuous boundaries. Furthermore, the measurement process of the proposed method does not require excessive post-processing operations, and the model belongs to a lightweight network, which is easy to deploy. Good results were achieved on the HC18 dataset of Grand-Challenge and 300 cases collected from hospitals. Compared with other mainstream segmentation networks such as U-Net, Res-U-Net, U-Net++, CE-Net, etc., the proposed method is with higher segmentation accuracy and smaller measurement error.
Key words: prenatal ultrasoundhead circumference measurementboundary segmentationconvolution neural networklightweight
胎儿头围是产前超声检查中最重要的生物学特征之一,它可以监测胎儿的生长发育情况,估计胎龄以及确定何种方式分娩等.根据产科超声指南[1],现有的胎儿头围测量方法几乎都是在2D胎儿丘脑标准平面上进行的.超声医生通常需要手动识别近似椭圆的颅骨边界,利用设备上的后处理软件勾勒椭圆并计算周长,从而测量出头围,如图 1所示.测量过程对超声医生来说既不方便又耗时, 由于颅骨边界的识别主要是由医生的经验及技术决定的,因此不同医生在同1张标准平面上测量出的头围会存在较大的观察者间误差[2],并且同1名医生的多次测量结果也会存在观察者内部误差,结果很难复现.近年来,计算机辅助检测已经广泛应用到产前超声筛查领域中,它可以减少观察者间误差,有效提高超声医生的工作效率.传统的机器学习方法需要先验知识人工设计特征,这种方式测量精度相对较低,鲁棒性较差[3-5].卷积神经网络在胎儿头围自动测量方面已经取得了一些成果[6-8].然而,这些方法仍然有一些局限性.首先,模型的输入需要1个非常严格的标准切面,即模型的输入需要拥有清晰且完整的颅骨边界,这时上述深度学习算法才可以获得相对较高的测量精度.在超声医生手动寻找标准平面过程中,由于受到邻近相似组织、羊水环境因素、医生操作经验等影响,标准切面往往会存在边界稍许模糊或者局部不完整的情况,不过这是可以接受的,经验丰富的医生可以根据对相关组织结构的了解,精准地勾勒出颅骨边缘,并不会影响头围测量的准确性.但是这种情况对于深度学习模型来说是一种极大的挑战.目前,凭借深度学习算法准确地测量头围,需要医生花费更多的时间获取“更严格”的标准平面.其次,直接分割颅骨边缘难度较大,精度较低.目前用于胎儿头围测量的深度学习方法基本上都是基于颅骨区域分割,再通过执行复杂的图像后处理操作进而计算出头围,如图 2所示.但是这些方法的网络结构往往比较复杂,模型参数与计算量较大,限制了模型部署的可能性.
图 1(Fig. 1)
 | 图 1 医生手动测量胎儿头围示意图Fig.1 Schematic diagram of fetal head circumference through doctors' manual measurement |
图 2(Fig. 2)
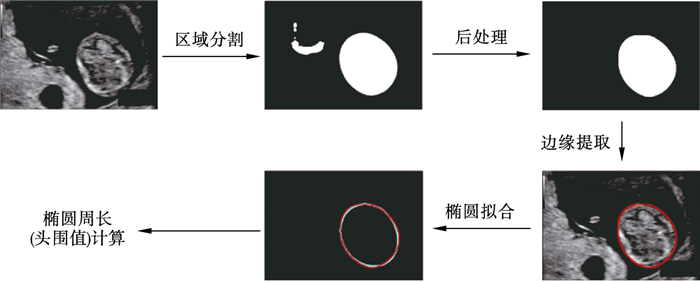 | 图 2 基于区域分割方法的胎儿测量流程示意图Fig.2 Schematic diagram of fetal measurement flow based on region segmentation method |
本文提出了一种新颖的双分支卷积神经网络直接分割颅骨边缘,并基于边缘分割结果直接进行椭圆拟合,从而获得椭圆参数及周长.在所提出的双分支卷积网络中,一个分支设计成标准的分割分支,另一个分支是获取可用作聚类的嵌入向量的分支,双分支结构通过共享层相互促进,可以分割出连续、光滑的颅骨边缘.区别于基于颅骨区域分割方法,本方法的测量过程无需过多的后处理操作就可以得出高精度的头围测量结果.与其他经典网络相比,本文方法在局部区域颅骨边缘不清晰或者不连续的标准切面上,仍然可以得到较好的分割结果,证明了该方法的强鲁棒性.此外,本文提出的网络结构属于轻量级网络,便于部署,具有很高的应用价值.
1 双分支卷积神经网络方法1.1 实验数据及其预处理实验数据由两部分组成,其中一部分数据来源于全球挑战赛Grand-Challenge中的HC18公共数据集[9],另一部分数据采集自中国医科大学附属盛京医院.HC18公共数据集包含1 334幅2D超声胎儿丘脑标准平面,旨在比较各种人工智能算法在此数据集上的测量精度.其中训练集999幅,测试集335幅,均包含早、中、晚妊娠期的标准切面,每幅图像的尺寸(单位:像素,下同)为800×540,像素范围从0.052 mm到0.326 mm.为了提高挑战难度,数据中存在一些颅骨边缘局部不清晰或者不连续的标准切面,用来验证算法的鲁棒性.300幅2D超声胎儿丘脑标准平面采集自中国医科大学附属盛京医院产科2015年至2018年超声影像数据库,数据由1名8年以上相关工作经验的超声医生进行手动标注,标注标准与HC18数据集相同,以近似颅骨边缘的椭圆为金标准,并且所有图像均对病人的隐私数据进行了处理.图像尺寸为704×576或640×480,像素范围从0.05 mm到0.25 mm.
将HC18数据集中的训练集999幅图像划分出600幅用作新的训练集,剩余的399幅作为验证集,测试集为HC18数据集中的测试集335幅图像及医院采集的300幅图像.将新的训练集进行数据增广,增广策略包括角度旋转、尺度变换、Gamma变换、随机翻转及其之间的组合.图像尺寸在输入到网络之前统一调整为400×272.数据预处理流程如图 3所示.为了与其他神经网络模型作对比,实验使用了两种分割掩码,一种掩码是分割颅骨边缘,见图 4b;另一种掩码是分割颅骨区域,见图 4c.
图 3(Fig. 3)
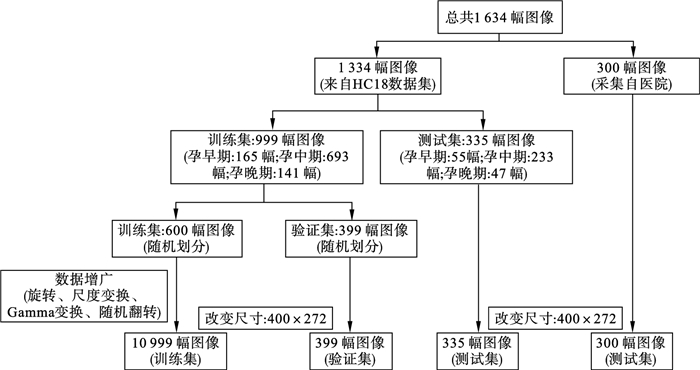 | 图 3 数据预处理流程图Fig.3 Flow chart of data preprocessing |
图 4(Fig. 4)
 | 图 4 2种掩码示意图Fig.4 Schematic diagram of two masks (a)—原图;(b)—分割颅骨边缘;(c)—分割颅骨区域. |
1.2 模型构建模型构建的动机是设计一个端到端的卷积神经网络直接分割颅骨边缘,同时尽量避免局部相似组织结构、人为因素对分割性能的干扰,兼具模型轻量化,便于部署.
本文提出一种双分支卷积神经网络,从总体架构上看是一个非对称的编码-解码结构,在解码部分分出2个分支,如图 5所示.为了降低模型的计算量与参数量,使得模型轻量化,本方法并没有像U-Net网络结构那样使用对称式的编码-解码结构及跨连接,本方法的编码部分是基于ERF-Net[10]改进的,主要由多个非对称空洞卷积单元组成.空洞卷积单元由两组3×1和1×3卷积组成,在第二组中使用空洞卷积,如图 6所示.空洞卷积有效地扩大了卷积视野,增加了全局信息,同时也为后续的边缘特征聚类提供了丰富的信息,对几乎占据图像全屏视野的孕晚期胎儿颅骨边缘长距离特征提取效果提升明显.下采样部分对输入使用3×3的卷积(步长为2)与最大池化操作,然后将2个结果堆叠(concat操作).为了使颅骨边缘分割得更加连续、光滑,在解码部分提出了一种双分支结构,一个分支用作分割,另一个分支用作获取可用作聚类的嵌入向量.上采样使用正常的反卷积.分割分支的输出为2×400×272,其中输出通道为2,代表两种类别,一类是颅骨边缘,一类是背景.聚类分支的输出为4×400×272,其中输出通道为4.嵌入向量分支的作用是获得颅骨边缘特征映射到4维特征空间中的嵌入向量,嵌入向量通过聚类损失函数聚类后,再通过空间坐标找到颅骨边缘相应位置上的特征点,使得颅骨边缘特征更加光滑、连续.最后将嵌入分支的结果与分割分支的结果进行逻辑“与”操作,再使用DBSCAN[11]聚类方法再次聚类,得到最终的颅骨边缘分割结果.通过OpenCV中的椭圆拟合函数fitEllipse直接对分割边缘结果进行拟合得到椭圆的关键参数,并基于文献[12]计算椭圆周长,即头围,计算公式如式(1)所示:
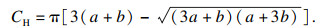 | (1) |
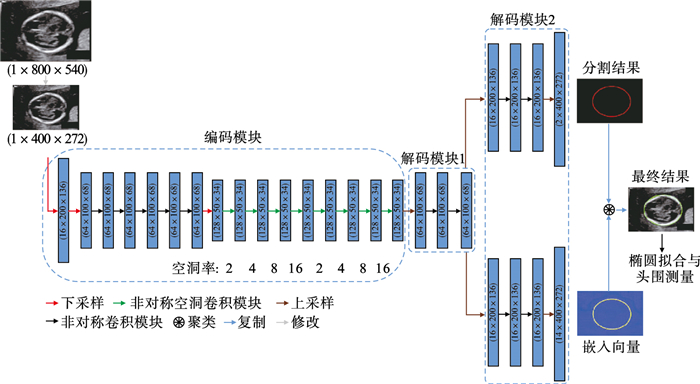 | 图 5 网络模型结构Fig.5 Network model structure |
图 6(Fig. 6)
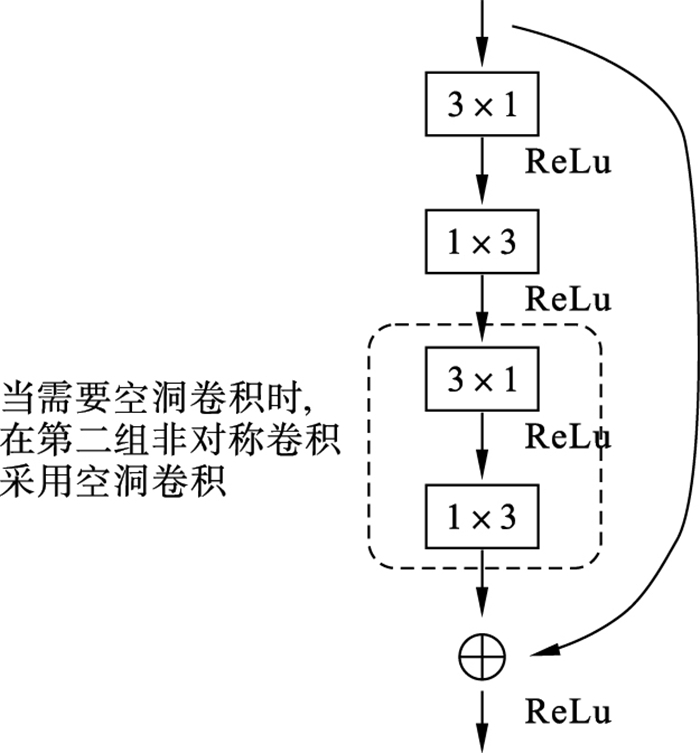 | 图 6 非对称卷积模块与空洞卷积模块Fig.6 Asymmetric convolution module and dilated convolution module |
其中:CH表示椭圆周长;a表示椭圆长轴长;b表示椭圆短轴长.
1.3 损失函数对于本文提出的双分支边缘分割模型,采用了二分类交叉熵损失(Lbce)、DICE损失(Ldice)和聚类损失(Lclu)[13]来约束网络训练.二分类交叉熵损失与DICE损失作用在分割分支,聚类损失作用在嵌入向量分支.总的损失函数可表示为
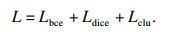 | (2) |
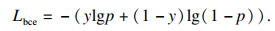 | (3) |
DICE损失函数可表示为
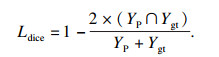 | (4) |
聚类损失函数可表示为
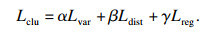 | (5) |
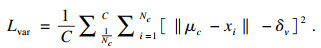 | (6) |
 | (7) |
Lreg表达式为
 | (8) |
2 实验与结果分析2.1 实验设置本文代码使用pytorch1.4框架,在2块NVIDIA GeForce RTX 2080TI显卡上进行训练.为了对比其他经典网络模型,使用颅骨边缘及颅骨区域两种掩码训练模型,将区域分割得到的结果经过后处理得到的最优结果进行对比.模型训练过程中使用的超参数如表 1所示.
表 1(Table 1)
| 表 1 超参数设置 Table 1 Hyperparameters setting | ||||||||||||||||
2.2 实验结果及分析本文提出的方法在Grand-Challenge中HC18数据集上取得较好的结果.在测试集(335幅图像)上,平均DICE得分为97.97,平均Hausdorff距离为1.21 mm,平均绝对误差为1.78 mm,平均误差为0.11 mm,与比赛第一名的各项指标对比如表 2所示.可以看出本文方法在分割精度上超越了HC18挑战赛的第一名,但是在最终的测量精度上略低于第一名,可能是由于本文并没有使用比较复杂的后处理来计算最终的头围结果.
表 2(Table 2)
| 表 2 本文方法与HC18挑战赛第一名对比 Table 2 Comparison between the method in this paper and the first place in HC18 challenge | ||||||||||||||||||||
为了更加全面比较模型的性能,本文方法与U-Net[14], Res-U-net[15], U-Net+ +[16], CE-Net[17]等其他经典分割网络在分割精度、测量精度、模型参数量、模型浮点运算量方面进行了对比,如表 3所示,其中以区域为掩码的分割结果均是经过后处理得到的最优结果.从表 3中可以看出,本文方法在分割精度、头围测量精度上均表现最佳,特别是颅骨边缘分割结果,精度甚至超过了其他经过复杂后处理所得到的结果,证明了所提方法的有效性.同时,本文方法的模型参数量、计算量也比较少,属于轻量化网络,便于部署.
表 3(Table 3)
| 表 3 不同模型结果对比 Table 3 Results comparison of different models | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
与其他模型分割结果的对比如图 7、图 8所示.图 7展示输入图像是具有连续、清晰颅骨边缘的对比结果,可以看出当输入图像是“严格”的标准切面时,分割结果相差不大.但是这种标准切面需要依赖超声医生的手法及经验,并且寻找过程相对耗时长一些,降低了医生的工作效率.图 8展示输入图像是具有局部不连续或者边缘不清晰颅骨边缘的对比结果.这种“非严格”的标准切面在临床上是经常会出现的,本文所提出的网络使用很多空洞卷积可以获得更多的全局信息,同时嵌入向量分支又可以对颅骨边缘特征进行聚类,间接修正颅骨边缘的分割结果,从而得到更加连续、光滑的边缘分割结果,凸显出本文模型较强的鲁棒性.
图 7(Fig. 7)
 | 图 7 不同模型分割结果对比(输入为“严格”标准切面)Fig.7 Segmentation results comparison of different models(input as "strict" standard plane) |
图 8(Fig. 8)
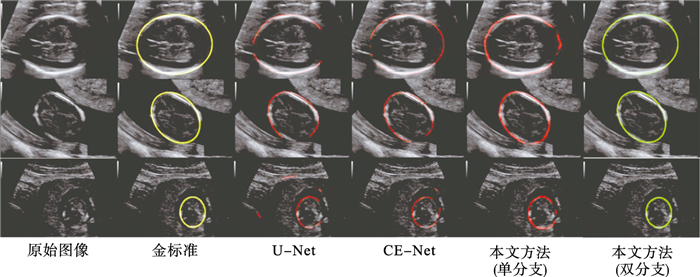 | 图 8 不同模型分割结果对比(输入为“非严格”标准切面)Fig.8 Segmentation results comparison of different models(input as "non-strict" standard plane) |
使用计算机辅助检查方法测量的头围是可复现的,并且可以辅助经验少的医生快速准确地测量胎儿头围,提高医生工作效率,同时可以避免不同医生间的测量误差以及医生自己重复测量的误差.为了体现观察者间的测量差异,从医院采集300幅标准切面,并由2名医生给出头围测量结果与本文方法进行比较,其中1名是具有8年以上超声工作经验的专家级医生,另1名医生从事超声工作刚满1年.以专家的测量结果为金标准,比较了专家、年轻医生自身多次测量的误差,年轻医生与专家之间的测量误差,本文所提出的模型与专家之间的测量误差,如表 4所示.可以看出专家和年轻医生均存在一定的自身重复测量误差,但误差并不大.但是,专家与年轻医生之间的测量误差较大,平均绝对误差为(2.21±1.12)mm,平均误差为(1.55±1.89)mm,说明年轻医生的经验及技术可能会影响到测量结果,与专家的测量结果有一定的差距.本文提出的模型与专家的测量结果平均绝对误差为(1.76±1.66)mm,平均误差为(-0.03±1.24)mm,明显小于年轻医生与专家之间的测量误差,说明本文方法与专家的测量结果相近,具有较高的测量精度.
表 4(Table 4)
| 表 4 观察者间的测量差异 Table 4 Measurement differences between observers | ||||||||||||||||||||
为了更直观地呈现本文方法的测量结果与金标准的一致性,在HC18的验证集上(399幅图像)绘制Bland-Altman图,如图 9所示.从图中可以看出,在95% 置信区间的平均误差为-0.03 mm,误差范围从-1.88 mm到1.82 mm.在置信区间外的点主要集中在孕早、中期,孕晚期几乎没有,从侧面证明孕晚期颅骨发育较好,边缘清晰,更有利于模型识别.从整体趋势上看,大部分点集中在置信区间内,且围绕在均值区域附近,可以推断模型的测量结果与金标准有较高的一致性.
图 9(Fig. 9)
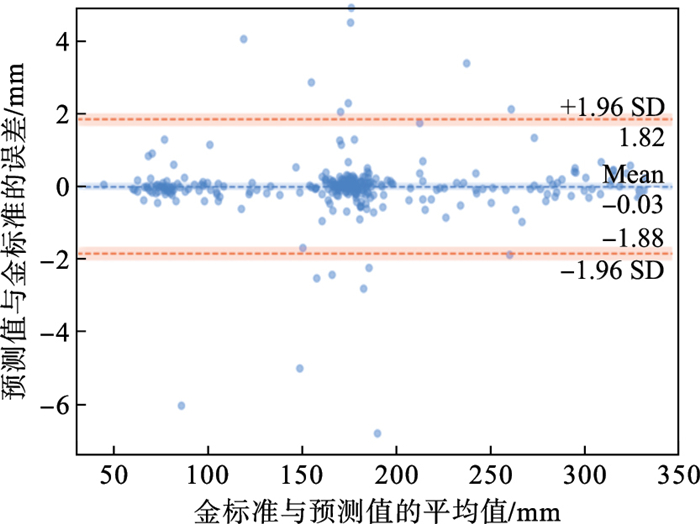 | 图 9 本文方法测量结果与金标准的Bland-Altman图Fig.9 The Bland-Altman plots of measurement results of the method in this paper and the gold standard |
3 结语本文提出了一种新颖的基于卷积神经网络的胎儿头围智能测量方法.本文方法可在2D超声丘脑标准平面上自动分割胎儿颅骨边缘并测量头围.区别于传统的区域分割方法,本文方法是一种端到端的胎儿颅骨边缘分割网络,并不需要复杂的后处理操作即可获得较高精度的胎儿头围测量值.即使在面对局部边缘不连续或者不清晰的标准切面时,本方法仍然可以获得较好的测量结果,具有较高的鲁棒性.通过大量的对比实验,验证了本文方法的有效性.此外,所提方法属于轻量化网络,便于部署,具有一定的商用价值.
参考文献
| [1] | Chudleigh T. The 18+0-20+6 weeks fetal anomaly scan national standards[J]. Ultrasound, 2010, 18(2): 92-98. DOI:10.1258/ult.2010.010014 |
| [2] | Sarris I, Ioannou C, Chamberlain P, et al. Intra-and interobserver variability in fetal ultrasound measurements[J]. Ultrasound in Obstetrics & Gynecology, 2012, 39(3): 266-273. |
| [3] | Nadiyah P, Rofiqah N, Firdaus Q, et al. Automatic detection of fetal head using haar cascade and fit ellipse[C]//2019 International Seminar on Intelligent Technology and Its Applications(ISITIA). Piscataway: IEEE, 2019: 320-324. |
| [4] | van den Heuvel T L A, Dagmar D B, de Korte C L, et al. Automated measurement of fetal head circumference using 2D ultrasound images[J]. PLoS ONE, 2018, 13(8): 0200412. |
| [5] | Zhang L, Dudley N J, Lambrou T, et al. Automatic image quality assessment and measurement of fetal head in two-dimensional ultrasound image[J]. Journal of Medical Imaging, 2017, 4(2): 024001. DOI:10.1117/1.JMI.4.2.024001 |
| [6] | Sobhaninia Z, Emami A, Karimi N, et al. Localization of fetal head in ultrasound images by multiscale view and deep neural networks[C]//The 25th International Computer Conference, Computer Society of Iran(CSICC). Piscataway: IEEE, 2020: 1-5. |
| [7] | Skeika E L, Luz M R, Fernandes B J T, et al. Convolutional neural network to detect and measure fetal skull circumference in ultrasound imaging[J]. IEEE Access, 2020, 8: 191519-191529. DOI:10.1109/ACCESS.2020.3032376 |
| [8] | Yang X, Wang X, Wang Y, et al. Hybrid attention for automatic segmentation of whole fetal head in prenatal ultrasound volumes[J]. Computer Methods and Programs in Biomedicine, 2020, 194: 105519. DOI:10.1016/j.cmpb.2020.105519 |
| [9] | Grand-Challenge HC18[EB/OL]. (2018-07-13)[2021-10-03]. https://hc18.grand-challenge.org, 2018. Z. |
| [10] | Romera E, Alvarez J M, Bergasa L M, et al. ERF-Net: efficient residual factorized convnet for real-time semantic segmentation[J]. IEEE Transactions on Intelligent Transportation Systems, 2018, 19(1): 263-272. DOI:10.1109/TITS.2017.2750080 |
| [11] | Birant D, Kut A. ST-DBSCAN: an algorithm for clustering spatial-temporal data[J]. Data & Knowledge Engineering, 2007, 60(1): 208-221. |
| [12] | Irene K, Aditya P, Haidi H, et al. Fetal head and abdomen measurement using convolutional neural network, hough transform, and difference of Gaussian revolved along elliptical path(Dogell)algorithm[EB/OL]. (2019-11-14)[2021-10-08]. https://arxiv.org/abs/1911.06298. |
| [13] | Brabandere B D, Neven D, Gool L V. Semantic instance segmentation for autonomous driving[C]//Computer Vision & Pattern Recognition Workshops. Piscataway: IEEE, 2017: 7-9. |
| [14] | Ronneberger O, Fischer P, Brox T. U-net: convolutional networks for biomedical image segmentation[C]//Medical Image Computing and Computer-Assisted Intervention(MICCAI). Berlin: Springer International Publishing, 2015: 234-241. |
| [15] | Xiao X, Shen L, Luo Z M, et al. Weighted Res-UNet for high-quality retina vessel segmentation[C]//2018 9th International Conference on Information Technology in Medicine and Education(ITME). Hangzhou, 2018: 327-331. |
| [16] | Zhou Z W, Siddiquee M M R, Tajbakhsh N, et al. UNet++: a nested U-net architecture for medical image segmentation[C]//The 4th Deep Learning in Medical Image Analysis(DLMIA) Workshop. Berlin: Springer International Publishing, 2018: 3-11. |
| [17] | Gu Z W, Cheng J, Fu H Z, et al. CE-Net: context encoder network for 2D medical image segmentation[J]. IEEE Transactions on Medical Imaging, 2019, 38(10): 2281-2292. DOI:10.1109/TMI.2019.2903562 |
| [18] | Badrinarayanan V, Kendall A, Cipolla R. Segnet: a deep convolutional encoder-decoder architecture for image segmentation[J]. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2017, 39(12): 2481-2495. DOI:10.1109/TPAMI.2016.2644615 |
| [19] | Qiao D H, Zulkernine F. Dilated squeeze-and-excitation U-net for fetal ultrasound image segmentation[C]//2020 IEEE Conference on Computational Intelligence in Bioinformatics and Computational Biology(CIBCB). Piscataway: IEEE, 2020: 1-7. |
