

1. 东北大学 信息科学与工程学院,辽宁 沈阳 110819;
2. 辽宁科技大学 电子与信息工程学院, 辽宁 鞍山 114051
收稿日期:2021-12-10
基金项目:国家自然科学基金资助项目(61871106);辽宁省重点研发计划项目(2020JH2/10100029)。
作者简介:魏颖(1968-),女,辽宁本溪人, 东北大学教授,博士生导师;
李伯群(1970-), 男,辽宁鞍山人,辽宁科技大学教授。
摘要:准确分割核磁共振(magnetic resonance,MR)图像中的脑组织是临床诊断、手术计划和辅助治疗的关键步骤.深度学习在各种图像分割任务中表现出巨大潜力,现有模型没有一种有效方法汇总远距离像素间的关系.在网络解码阶段不能很好地融合不同层级的特征,导致无法准确定位.为克服上述问题, 本文提出一种基于空间自注意力机制和深度特征重建的脑MR图像分割方法,构建了一个可以融合3维信息的2D模型,可快速准确对3D结构图像进行密集预测.在MRBrainS13数据集和IBSR数据集上进行充分地实验研究,结果表明本文方法在3D多模态和单模态脑MR图像分割方面优于目前的2D模型,运算和推理时间相比3D模型小很多,性能却十分接近.
关键词:脑图像分割全卷积网络空间自注意力通道注意力深度特征重建
Brain MR Image Segmentation Based on Spatial Self-attention Mechanism and Depth Feature Reconstruction
WEI Ying1, LIN Zi-han1, QI Lin1, LI Bo-qun2


1. School of Information Science & Engineering, Northeastern University, Shenyang 110819, China;
2. School of Electronic & Information Engineering, University of Science and Technology Liaoning, Anshan 114051, China
Corresponding author: LI Bo-qun, lbqhylyxab@163.com.
Abstract: Accurate segmentation of brain tissue in MR images is a key step in clinical diagnosis, surgical planning and adjuvant treatment. Deep-learning shows great potential in various image segmentation tasks, and existing models do not have an effective way to summarize the relationship between long-distance pixels. In the network decoding stage, the features of different levels cannot be well integrated, resulting in the inability to accurately locate. To overcome the above problems, this paper proposes a brain MR image segmentation method based on spatial self-attention mechanism and depth feature reconstruction, and constructs a 2D model that can fuse 3D information, which can quickly and accurately perform dense prediction on 3D structural images. The proposed method is fully experimented on MRBrainS13 data sets and IBSR data sets, and the results show that the model outperforms the current 2D model in 3D multimodal and unimodal brain MR image segmentation, with less computing and inference time compared to the 3D model, whereas the performance is very close.
Key words: brain image segmentationfully convolutional networkspatial self-attentionchannel attentiondepth feature reconstruction
核磁共振成像(magnetic resonance imaging, MRI)可以拍摄出高对比度、高分辨率的软组织空间图像,是进行结构性脑分析的重要方式.脑MRI图像的定量分析已被广泛用于脑部疾病的研究中.例如,在诊断阿尔茨海默病、癫痫、精神分裂症等神经疾病[1]时,需要对脑组织进行核磁共振成像,并对MRI图像进行分割和测量.但手动分割完成上述功能时,会非常耗时并需要高度的专业知识,因此,自动脑组织分割将极大地帮助医疗诊断和制定治疗计划,是当前研究的热点[2].
医学图像具有较高的复杂性且缺少简单的线性特征,其摄取方式的独特性为医学图像的精确分割带来了许多难点与问题:医学图像的特殊性会使医学图像存在较多的噪声及容积效应,造成组织间边界模糊、不明确等问题,这为医学图像分割提升了难度;医学图像数据需要耗费大量的人工进行标签标注,标注数据的稀缺性一直是分割面临的问题;人体组织结构复杂,不同结构间紧密相连,边缘灰度差异不明显,体积较小的结构边缘难以准确分割,这为医学图像分割提高了难度.
以卷积神经网络为代表的深度模型被广泛引入脑MRI图像分割研究中[3-4].基于网络体系结构,深度学习类分割方法大致可分为两类:基于图像块的方法[5]和基于语义分割的方法[6-8].基于图像块的方法使用中心像素附近邻域的局部图块作为输入,在一张图片上裁剪出大量图像块,汇总所有图像块的分类信息,得到整体图像像素的密集预测.Moeskops等[5]使用多尺度的2D CNN,构建3个单独的支路网络,并在预测之前组合了这些特性.这种多尺度设计使得网络能够获得细粒度的局部和全局信息,以更好地对目标像素进行分类[5].Lyksborg等[9]利用轴面、矢状面和冠状面的二维切片分别训练了3个网络.与文献[5]不同,此方法中3个网络的集成是在预测步骤之后进行的,即基于多数投票的方法对3个网络的结果进行汇总.
为了更好地保留图像空间信息,Long等[10]将全卷积神经网络(fully convolutional networks, FCN)引入图像分割,提出基于语义分割的方法,该方法极大地提升了分割的准确率,使全卷积结构成为深度学习语义分割算法的基本结构.Ronneberger等[11]在FCN的基础上进行进一步改进,提出了一种编码-解码结构,称为U-Net.U-Net在分割医学图像方面取得了巨大成功,即使有标记的训练数据很少,也可以产生很好的分割效果,一定程度上,U-Net已经成为医学图像分割的基准.
基于语义分割方法在分割效率和发展前景上更占优势,一些局限性阻碍了这种方法的有效性.卷积神经网络可以提升高层次特征的抽象表示能力,但高层特征无法充分获取空间信息,在一定程度上降低了医学图像分割的定位能力.脑部不同组织间边缘像素的定位往往十分困难[12],卷积神经网络方法使用卷积等局部操作捕获特征,重复使用局部操作导致很难对远距离像素间的关系进行建模,使网络性能无法进一步提升.解码阶段只有相邻层级的特征通过通道串联或加和的方式进行结果预测,无法很好地对不同层级的特征进行融合.针对上述问题,在U-Net模型基础上设计了两个基于注意力机制的模块,并拓展了网络的解码结构,提出了一种注意力深度特征重建网络(attentive deep feature reconstruction network)来分割脑MR图像.
该网络使用了3种模态的脑部图像作为输入,通过不同模态间的对比,提升了不同脑组织间的分割精度.在实际应用中,没有为不同的模态分配独立的参数,而是3种模态共用同一组主干网络,实验表明这既节省了大量参数,又使主干网络得到充分的训练.
在使用3模态图像的基础上,本文又在如下3个方面对全卷积神经网络的方法进行了改进:
1) 对脑MRI扫描的二维切片进行一种特殊的数据增强,堆叠连续3个2D切片,构造伪RGB形式的图像.
2) 使用空间自注意力方法对原始U-Net网络进行改进,空间自注意力使网络能够直接计算任意两个位置之间的交互关系,捕获远程依赖性,更好理解脑部组织结构并准确预测.
3) 提出了1个深度特征重建模块,使用具有空间上下文信息的深度特征对不同层级的特征进行重新加权.提升重要特征,抑制不重要的特征,同时也让不同层级的特征更具有一致性.
1 注意力深度特征重建网络深度学习语义分割领域的基础工作是全卷积神经网络FCN.该方法的出色之处在于它使用分类卷积神经网络作为其子模块以产生层次化的特征,然后组合提取到的特征对图像做出像素级别的预测.
在医学图像分割领域,大多数分割算法均是U-Net网络的改进[13-14].这些网络由对称的编码网络和解码网络两部分构成.在编码阶段,使用去掉全连接层的分类网络作为分割的编码器,以产生低分辨率的图像表示或者特征映射送入解码器.该阶段的图像分割问题在于如何学习到足够优秀的特征对图像成功进行编码.解码部分一般由一系列的上采样操作即反卷积层和反池化层组成,然后接上1个softmax分类器预测像素级别的标签.
除了解码器的结构设计,U-Net架构中最巧妙的是引入跳跃连接.在4个级别的特征图中,对编码器的特征进行池化之前,编码端卷积层的输出被转移到解码器.将这些特征与上采样后的特征进行通道串连,并将连接的特征映射继续解码并上采样到分辨率更高的层级.这些跳跃连接使得网络可以找回编码端池化操作丢失的空间信息,更好地融合语义信息与位置信息.
原始U-Net对空间上下文信息捕获相对匮乏,即忽略了图像中不同位置的空间相关性.在医学图像中,不同组织结构相互依赖,空间上下文信息十分重要,仅通过通道串联融合不同阶段的特征无法处理不同阶段特征之间的差异性,重复进行特征融合导致重要特征被忽略, 本文提出注意力深度特征重建网络分割脑MR图像.详细介绍网络中包含的两个模块: 自注意力模块和深度特征重建模块.自注意力模块在空间维度上捕获远程上下文信息,深度特征重建模块可以用深度特征中的语义信息来指导低层次特征进行上采样重建.本文将二者聚合以进一步改进分割结果.
1.1 网络概观网络的整体架构如图 1所示,网络的输入为三模态图像,整体架构采用编码-解码结构的方式.编码结构经过多次下采样,旨在提取图像的上下文语义信息.因医学图像具有边界模糊等特点,精准分割需要依赖低级特征和高级语义特征,因此整体模型通过跳跃连接将高分辨率与低分辨率信息进行融合,得到最终的分割结果.
图 1(Fig. 1)
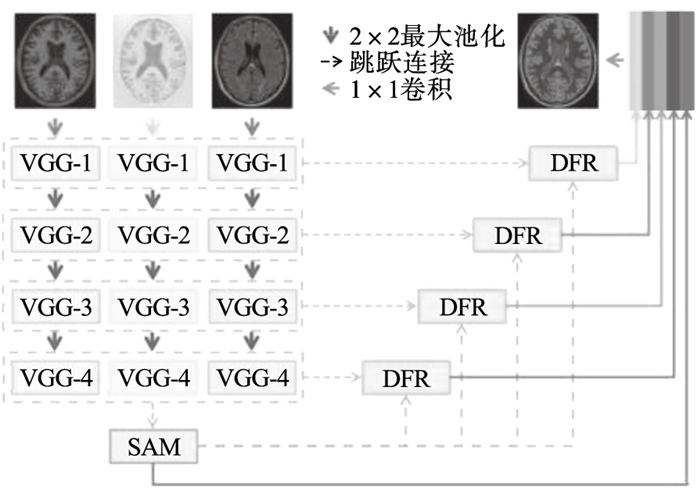 | 图 1 网络结构示意图Fig.1 Illustration of network architecture |
将VGG-16网络的前10层作为编码器网络,根据VGG-16网络前10层的特征图大小,将其分成了4个层级,分别对应图 1中的VGG-1, VGG-12, VGG-3, VGG-4.网络移除VGG-16网络末端的全连接层和最后的三组卷积层,最终特征映射的大小为输入图像的1/8.这样可以保留更多的细节,而无需添加额外参数.为了更好地在空间维度上捕获远程上下文信息,将来自编码网络的特征送入空间自注意力模块(spatial self-attention module, SAM),将空间自注意力模块SAM修正后的高级特征和各层级的特征同时送入深度特征重建模块(deep feature reconstruction module, DFR),自适应地对不同尺度的特征图进行上采样重建.将5个层级的特征融合进行密集预测得到最终的分割结果.
1.2 空间自注意力模块(SAM)上下文关系对于理解图像至关重要,尤其是图像中的一些远距离信息.传统FCN通过短距离卷积操作获得图像的局部感受野,忽略了图像中不同位置的空间相关性,易导致错误的分割结果.为了聚合空间上下文信息以增强特征表示,许多****做出了大量努力.Chen等[15]提出ASPP模块,使用不同空洞率的空洞卷积捕获上下文信息.基于空洞卷积方法只能收集少数周围像素的信息,不能生成密集的上下文信息.基于池化方法以非自适应的方式聚合上下文信息,并且所有像素都会采用相同的上下文信息,不能满足不同像素需要不同上下文的要求.Francesco等[16]利用RNN捕获远程上下文依赖信息,但这些方法只能隐含地捕捉全局关系,其有效性在很大程度上依赖于长期记忆的学习结果,计算低效.条件随机场(conditional random field,CRF)和马尔可夫随机场(Markov random field,MRF)被用于捕获远程依赖性,但这类基于概率图模型的工具无法很好融入卷积神经网络,不能端到端进行优化,往往仅用作后处理.
为丰富特征中的空间上下文信息,引入一个自注意力模块.首先计算某位置与所有位置之间的成对关系形成的注意力掩模,然后对所有位置的注意力掩模计算加权和,再与原始特征逐点相加形成最终输出.将详细说明如何通过自注意力模块聚合空间上下文信息.
空间自注意力模块如图 2所示.
图 2(Fig. 2)
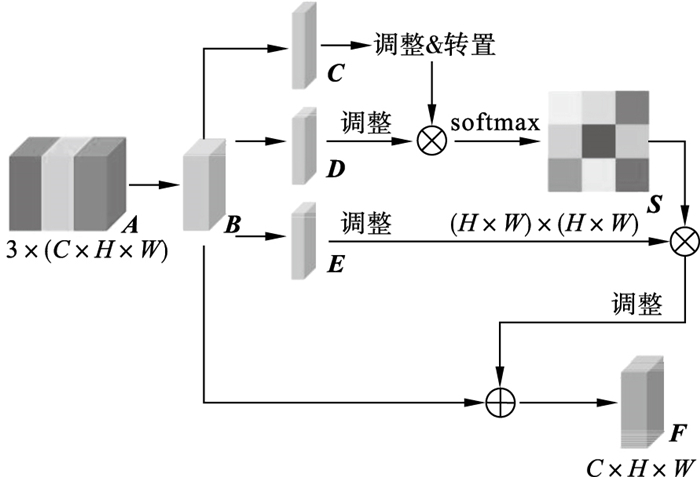 | 图 2 空间自注意力模块Fig.2 Spatial self-attention module |
给定3个模态的局部特征A∈RC×H×W,对其进行通道串联和1×1卷积通道降维,得到局部特征B∈RC×H×W,然后将其送入具有批归一化BN层和ReLU层的卷积层,分别生成两个新的特征映射C和D,其中{C, D}∈ RC×H×W.然后将其形状调整为RC×N,其中,N=H×W,代表特征的数量.在C和DT之间执行矩阵乘法,并应用一个softmax层计算空间注意力S∈ RN×N:
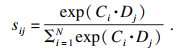 | (1) |
将特征B送入具有批归一化BN层和ReLU层的卷积层,以生成新的特征映射E∈ RC×H×W,并将其调整为RC×N.在E和ST之间进行矩阵相乘,并将结果形状调整为RC×H×W.将结果乘以一个尺度参数α,并和特征B进行元素相加,得到最终输出F∈RC×H×W:
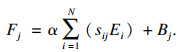 | (2) |
1.3 深度特征重建模块(DFR)在医学影像领域的分割模型中,多数方法使用类U型网络的编码-解码结构,并利用不同阶段的特征辅助预测.这种网络结构利用了不同层级的多尺度特征,通过通道串联融合不同阶段的特征.不同阶段的特征具有不同程度的判别性,这种操作无法处理不同阶段之间的差异性,由于缺乏具有强一致性的全局信息,容易导致预测不一致性.
重复进行特征融合往往会导致某些重要特征被忽略.在原始的卷积操作中,隐含的认为所有通道的权重是相等的,无法强调不同通道的差异.为了获得类内一致的预测,应该增强不同阶段特征的一致性,并抑制不具判别性的特征.
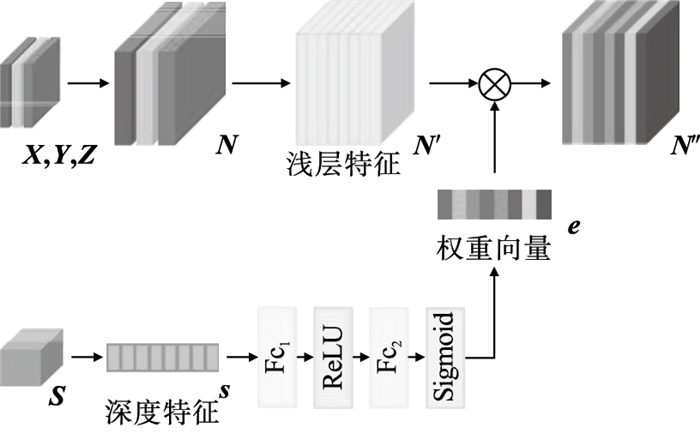
为了增强解码阶段不同层级特征的一致性,设计了一个基于通道注意力的深度特征重建模块,如图 3所示.该模块聚合包含全局上下文信息的高级语义特征和不同低级语义的特征,使空间自注意力修正后的特征计算通道注意力向量,对不同层级特征的重要性进行重建,使各个层级的特征更具有代表性和一致性.
图 3(Fig. 3)
 | 图 3 深度特征重建模块Fig.3 Deep feature reconstruction module |
在图 3中,蓝色块表示由空间自注意力SAM修正后的高级特征,黄色块代表不同层级的低级特征.对空间自注意力修正后的特征图S∈RN×A×B进行全局平均池化,得到一组深度特征向量s=[s1, s2, ?, sn]∈ RN×1×1, 对其进行式(3)操作以获得各个通道的代表值.
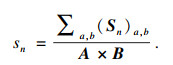 | (3) |
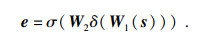 | (4) |
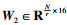
对于编码网络同一阶段的3个模态图像对应的特征为{X, Y, Z}∈RN×A×B,对其进行通道并联和上采样至原图像大小的操作,形成不同层级的三模态特征N∈ RN×α·A×α·B:
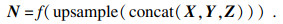 | (5) |
由于这种通道注意力操作能够编码不同特征图之间的依赖性,使深度特征重建模块可以更改不同阶段特征的权重.因为深度特征重建模块采用高级语义特征对各层级特征进行辅助指导,使浅层网络可以学习到更具区分能力的特征,避免学习到对分割无益的无关特征,从而达到增强特征一致性的作用,极大改善了网络性能.
2 实验与分析2.1 数据集和预处理在MRBrainS13和IBSR18[17]数据库上评估了本文方法.MRBrainS13数据集包含不同程度萎缩和病变的患者的脑部扫描图像,在此数据集对有病变的图像进行评估.IBSR18数据集包含正常个体的脑部扫描图像,在此数据集上可以和更广泛的方法进行对比.MRBrainS13数据集提供了3T扫描数据,IBSR18数据集只提供了T1w扫描数据,因此在使用MRBrainS13和IBSR18数据集时,编码网络分别采用三模态输入和单模态输入,证明SAM和DRF在不同模态输入时的有效性.
2.1.1 MRBrainSMRBrainS13挑战的任务是将大脑MRI扫描分割为4种脑结构,即背景、脑脊液(CSF)、灰质(GM)和白质(WM).这些数据是在糖尿病患者和相应的有不同程度的脑萎缩和白质病变的对照组中获得的,是由荷兰乌得勒支大学医学中心提供.每个受试者提供多序列3T MRI脑扫描,包括T1加权、T1-IR和T2-FLAIR,数据尺寸为256×256×48.训练数据集由5个手动进行分割的样本组成.测试数据包括15个样本,仅提供原始扫描图像,不提供标签图像,在测评时将分割结果进行提交,由主办方独立进行分割准确性评估.
2.1.2 IBSR18IBSR18数据库包含18个正常受试者的脑组织切片数据.每个受试者提供一个T1w序列和对应的手动分割标签.MRI图像和手工分割标签是由马萨诸塞州总医院的形态计量分析中心提供的,尺寸为256 mm×256 mm×128 mm,数据集已进行了头骨剥离和偏移场矫正.
2.1.3 数据预处理在预处理步骤中,对原始二维切片图像使用了缩放和随机旋转的数据增强策略,并且还减去了训练图像中每个通道的平均值.本文使用了一种堆叠连续2D切片,构造伪RGB形式图像的数据处理方式.为了能够使用VGG网络第一层的预训练权重,需要人工构造1个3通道的伪RGB图像.为此,将脑部MRI扫描的n-1,n,n+1这3个切片作为RGB图像的3个通道,如图 4所示.
图 4(Fig. 4)
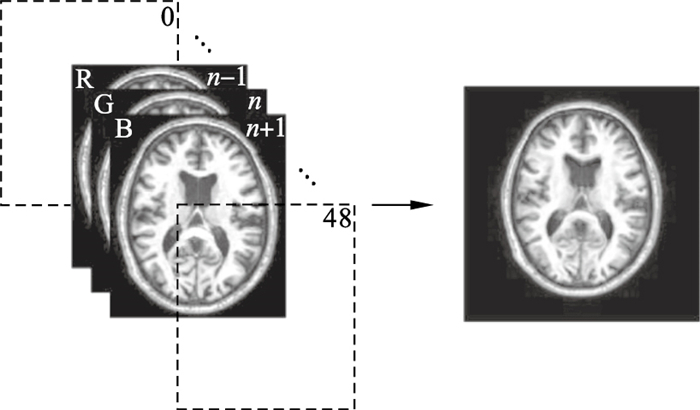 | 图 4 堆叠连续切片步骤Fig.4 Step of stack continuous slice |
这样做有两个好处:1)构造的每个2D图像都包含了一些3D的信息,同时使用2D网络进行分割又避免了3D FCN高昂的计算和内存要求;2)由于预训练权重都是使用自然场景下进行图像训练,构造2D的3通道RGB图像更适合使用有预训练权重的网络进行迁移学习.进行堆叠之后,再对3通道图像进行随机旋转和裁剪.
2.2 评估和比较MRBrainS13数据集的评估结果来自于赛事主办方,指标有3项:分别是每种组织类型(即GM,WM和CSF)分割的Dice系数(DC),Hausdorff距离的第95个百分位数(HD-95)和绝对体积差异(AVD).在IBSR18数据集中,只比较DC,因为很多方法只使用了这个评估指标.
Dice系数(DC)是医学影像分割评价指标中最广泛应用的一种度量方法,DC的计算式为
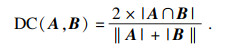 | (6) |
HD是对预测图形的形状相似性的一种度量,由于Dice指标的数值高低主要取决于图形的内部,对边界的刻画相对不敏感,使用HD进行评估能够对Dice做出较好的补充.HD的计算式为
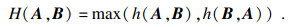 | (7) |
AVD为度量两个体积的绝对差异:
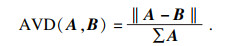 | (8) |
2.3 与其他方法比较2.3.1 MRBrain数据集本文方法与其他几种方法的结果对比如表 1所示.比较方法大致分为3类:分别是现有软件的分割结果FreeSurfer和FSL_Seg;基于图谱和机器学习的方法Random Forest[17]和Multi-atlas[18];基于深度学习的方法VoxResNet[6], MDGRU[19], PyraMiD-LSTM[20]和MixNet.
表 1(Table 1)
| 表 1 MRBrainS13不同方法结果对比 Table 1 Comparison of results of different methods of MRBrainS13 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
由表 1可知,分割软件在WM和GM的分割上接近多图谱方法Multi-atlas[18],但在CSF的分割上多图谱方法大幅度优于软件分割结果.基于随机森林方法[17]整体指标上十分接近深度学习类方法,这可能得益于作者在随机森林预测后又添加了条件随机场CRF进行后处理修正.但此方法推理时间过长,对单个样本进行预测,平均需要25 min,在对比方法中推理时间仅次于FreeSurfer.MRBrainS13测试集的平均结果如表 2所示.
表 2(Table 2)
| 表 2 MRBrainS13测试集结果 Table 2 MRBrainS13 test set results |
2.3.2 IBSR18数据集表 3为本文方法与其他几种方法的结果对比.脑脊液由脑室脑脊液和脑外沟回脑脊液两部分组成,在IBSR数据集进行实验的分割算法将脑沟回的脑脊液认为是灰质,在评估时会删除沟回脑脊液的结果,不将其纳入整体评估,这影响了算法的准确性.Valverde等[21]在考虑沟回脑脊液和不考虑沟回脑脊液的条件下重新评估了一些知名的有效方法.本文方法与Valverde评估方法保持一致.从表格中可以看出,本文方法较传统方法和深度学习类方法均有提升.
表 3(Table 3)
| 表 3 IBSR18不同方法结果对比 Table 3 Comparison of results of different methods in IBSR18 |
2.4 对比实验为了验证本文方法中各个部分的有效性,在MRBrainS13训练数据集上进行多个对比实验.本文方法在验证集上的分割结果如图 5所示.
图 5(Fig. 5)
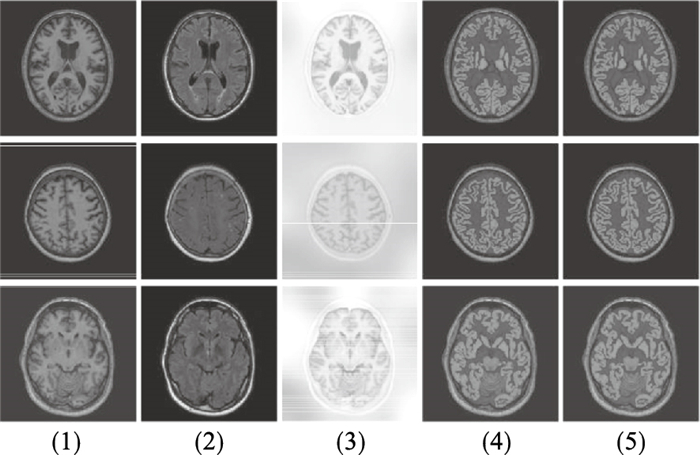 | 图 5 MRBrainS13验证集结果Fig.5 MRBrainS13 validation set results (1)—T1;(2)—T2-FLAIR;(3)—T1-IR;(4)—Label;(5)—算法结果. |
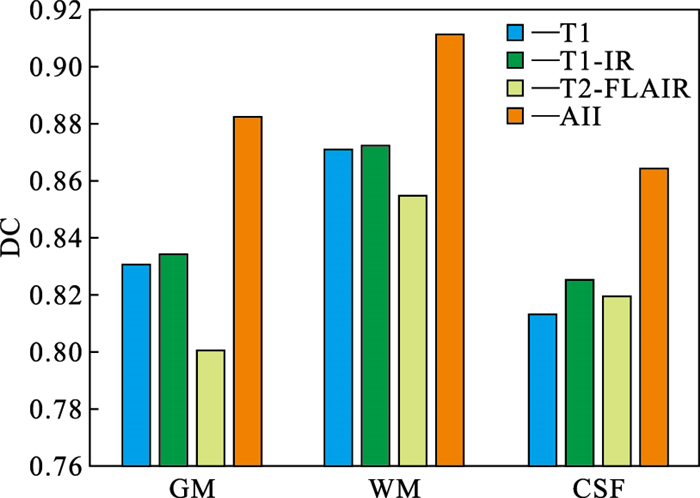
2.4.1 多模态图像有效性验证为了研究使用多模态数据的有效性,对验证数据进行了对比实验,其中DC指标的结果如图 6所示.
图 6(Fig. 6)
 | 图 6 不同模态图像的分割结果Fig.6 Segmentation results of images with different modalities |
由图 5可知,T1图像和T1-IR图像中脑组织间对比度更高,更有利于脑间不同组织的分割,T2-FLAIR图像中脑组织间对比度较低.由图 6可知,单独使用T2-FLAIR模态图像进行分割结果最差,单独使用T1图像和T1-IR图像进行分割结果相近.T1图像中沟回部分的脑脊液与颅骨之间对比度较弱,导致使用T1图像时CSF的分割效果并不十分理想,如图 5所示.T1-IR图像中脑组织与非脑组织之间差异大,T2-FLAIR中颅骨轮廓十分清晰,由图 6可知,单独使用T1-IR和T2-FLAIR图像进行分割时,CSF的分割结果均优于单独使用T1图像.这种差异体现了不同模态之间的互补特性,一定程度上解释了为何使用多模态图像的信息可以显著提高分割性能.所以,从实验结果的定量分析和实际观察的定性分析中均可证明使用多模态图像的有效性.
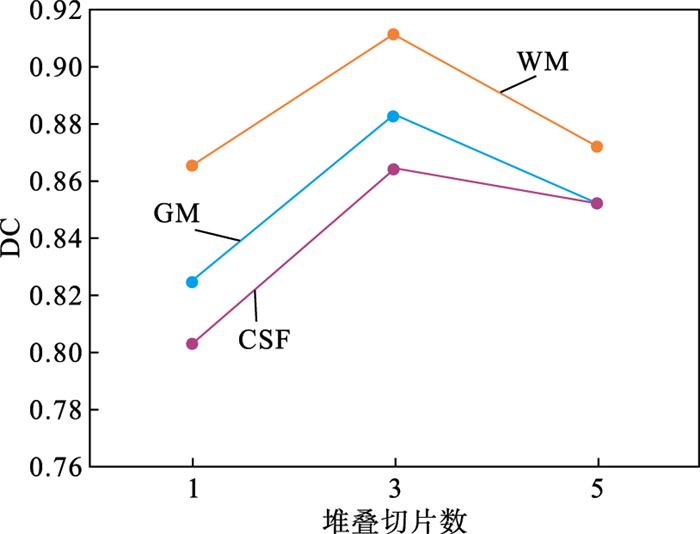
2.4.2 堆叠图像切片有效性验证为了研究数据预处理阶段堆叠切片操作的有效性,使用验证数据进行了对比实验,其中DC指标和推理时间的结果如图 7所示.
图 7(Fig. 7)
 | 图 7 堆叠图像切片分割结果Fig.7 Stacked image slice segmentation results |
由图 7中的折线图部分可知,堆叠切片数量为3时效果最好,不进行堆叠直接使用单个切片效果最差.只使用单个切片时,网络为完全的2D网络,无法获取第三个维度的空间信息,对于三维的脑部组织无法很好地进行预测.当堆叠三个相邻切片时,将一部分空间信息送入网络,使网络可以更好地做出预测.当堆叠5个相邻切片时,虽然引入了更多空间信息,但网络分割效果不仅没有提升反而比堆叠3个相邻切片变差了.这是由于堆叠5个切片无法使用预训练VGG网络的第一层权重,导致网络无法更好地进行迁移学习.另一方面也说明了空间信息并不是越多越好,距离相近的切片之间可能包含更多有利于网络做出预测的空间信息,而距离较远的切片之间差异往往较大,有可能会阻碍网络的预测.
2.4.3 多模态分支设计验证为了研究不同模态数据应该如何分配编码网络,本文使用验证数据进行对比实验,当3个模态图像作为输入,使用同一组编码网络和分别使用三组编码网络的最终分割结果十分相近.编码网络数量为1,3时,p分别为11.7,26.5 MB.3种模态图像使用同一组编码网络时网络参数量明显减少,耗费的计算资源更少.
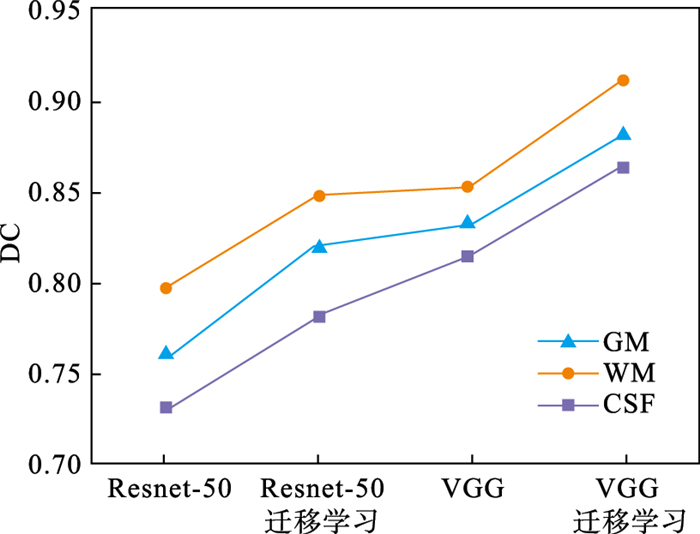
2.4.4 主干网络设置验证选取深度神经网络ResNet50和层数较浅的VGG网络进行对比.图 8中“ResNet-50”和“VGG”代表仅使用常规策略进行网络参数初始化.而两种网络标注“迁移学习”代表使用在IMAGENET大型数据集上预训练好的权重对网络进行参数初始化.由于医学影像类数据集规模较小,在脑部数据集上,较浅的网络比深层网络表现要更好.这可能是由于较少的数据无法让较深的网络得到充分训练.可知,预训练过的模型,对比没有预训练的模型,在分割结果上有很大提升.这也说明迁移学习的方式对规模较小的医学图像来说是十分有效, 如图 8所示.
图 8(Fig. 8)
 | 图 8 主干网络设置验证Fig.8 Trunk network Settings validation |
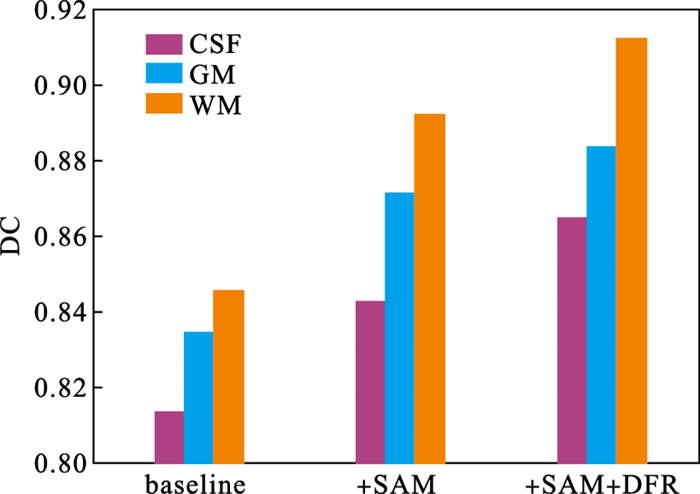
2.4.5 SAM和DFR模块有效性验证为验证提出的空间注意力模块和深度特征重建模块的有效性,使用MRBrainS13验证数据进行了对比实验,结果如图 9所示.
图 9(Fig. 9)
 | 图 9 SAM和DFR模块有效性验证Fig.9 SAM and DFR module validity verification |
使用3个模态图像作为输入的U-Net基线模型.由图 9可知,使用空间自注意力模块SAM后网络分割结果得到了有效提升,平均DC值可提升大约4 %,表明空间自注意力模块可以有效对上下文信息进行聚合.针对脑MR图像中丰富的先验信息,不同组织间相互依赖,远距离像素间进行信息传递可以有效提升分割精度.
在使用SAM模块的基础上使用深度特征重建模块DFR,进一步提升了网络性能,平均DC值有2 % 左右的提升,表明深度特征重建模块可以对各层级语义进行辅助指导,增强网络学习到的分割特征的判别性,进而改善网络的分割性能.
两种模型3种脑组织的分割结果如图 10所示,图中金标准为医生标注的分割结果即真实标签.由图 10可知本文方法能够更好地判断脑部组织的类别,尤其是脑部组织狭长细小的区域,如图 9中第2列的CSF图像和第4列的WM图像中黄色圈出的部分.对于组织边缘的位置能够作出更准确的定位,尤其是组织之间的细小区域,如图 9中第3列GM图像中黄色圈出的部分.
图 10(Fig. 10)
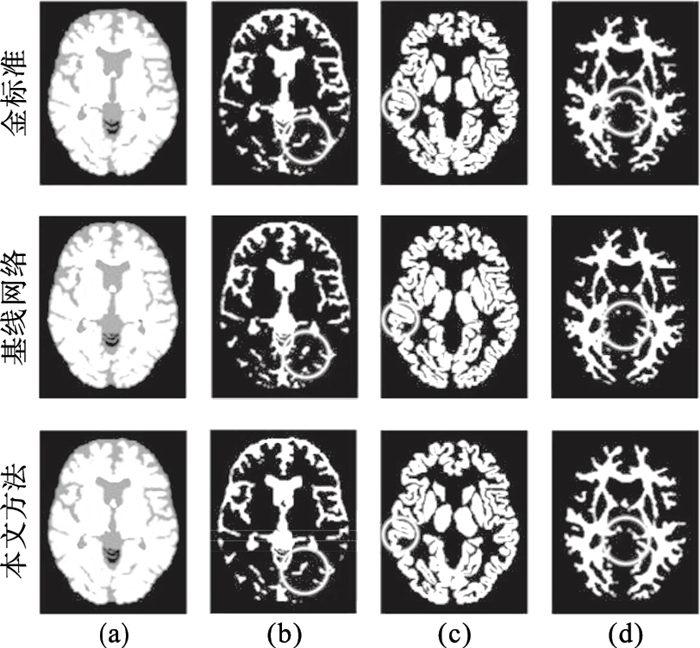 | 图 10 脑组织分割结果对比Fig.10 Comparison of brain tissue segmentation results (a)—原始图像;(b)—CSF;(c)—GM;(d)—WM. |
结果表明,针对脑MR图像,本文提出的空间自注意力模块SAM可以对长距离像素间进行信息传递,有选择性的聚合上下文信息,深度特征重建模块DFR可以指导网络学习到更有判别力的特征,针对脑MR图像中狭长、细小及边缘部分的组织分割有较好的提升效果.
3 结论对医学多模态数据进行特征提取,并在网络解码端进行特征融合.使用堆叠图像切片的方式对图像进行数据增强,将3维信息编码到图像的3个通道中,构建出具有空间信息的伪RGB形式图像,能够更好地挖掘样本数据的信息并进行迁移学习.实验证明了使用多模态数据和堆叠图像切片的有效性.得益于这两种数据的增强手段,本文仅使用2D网络便获得了比肩3D网络的效果,而且非常节省时间和计算量.
参考文献
| [1] | Nuzziello N, Ciaccia L, Liguori M. Precision medicine in neurodegenerative diseases: some promising tips coming from the microRNAs' world[J]. Cells, 2020, 9(1). DOI:10.3390/cells9010075 |
| [2] | Akkus Z, Galimzianova A, Hoogi A, et al. Deep learning for brain MRI segmentation: state of the art and future directions[J]. Journal of Digital Imaging, 2017, 30(4): 449-459. DOI:10.1007/s10278-017-9983-4 |
| [3] | Minaee S, Boykov Y Y, Porikli F, et al. Image segmentation using deep learning: a survey[J]. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2022, 44(7): 523-3542. |
| [4] | Devunooru S, Alsadoon A, Chandana P W C, et al. Deep learning neural networks for medical image segmentation of brain tumours for diagnosis: a recent review and taxonomy[J]. Journal of Ambient Intelligence and Humanized Computing, 2021, 12(1): 455-483. DOI:10.1007/s12652-020-01998-w |
| [5] | Moeskops P, Viergever M A, Mendrik A M, et al. Automatic segmentation of MR brain images with a convolutional neural network[J]. IEEE Transactions on Medical Imaging, 2016, 35(5): 1252-1261. DOI:10.1109/TMI.2016.2548501 |
| [6] | Chen H, Dou Q, Yu L, et al. VoxResNet: deep voxelwise residual networks for brain segmentation from 3D MR images[J]. NeuroImage, 2018, 170: 446-455. DOI:10.1016/j.neuroimage.2017.04.041 |
| [7] | Sun K, Xiao B, Liu D, et al. Deep high-resolution representation learning for human pose estimation[C]//Proceedings of the IEEE/CVF Conference on Computer Vision and Pattern Recognition. Long Beach, 2019: 5693-5703. |
| [8] | Gu R, Wang G, Song T, et al. CA-Net: comprehensive attention convolutional neural networks for explainable medical image segmentation[J]. IEEE Transactions on Medical Imaging, 2020, 40(2): 699-711. |
| [9] | Lyksborg M, Puonti O, Agn M, et al. An ensemble of 2D convolutional neural networks for tumor segmentation[C]// Scandinavian Conference on Image Analysis. New York: Springer, 2015: 201-211. |
| [10] | Long J, Shelhamer E, Darrell T. Fully convolutional networks for semantic segmentation[J]. IEEE Transactions on Pattern Analysis & Machine Intelligence, 2017, 39(4): 640-651. |
| [11] | Ronneberger O, Fischer P, Brox T. U-Net: convolutional networks for biomedical image segmentation[C]//International Conference on Medical Image Computing & Computer-assisted Intervention. Munich, 2015. DOI: 10.1007/978-3-319-24574-4_28. |
| [12] | Dolz J, Desrosiers C, Ayed I B. 3D fully convolutional networks for subcortical segmentation in MRI: a large-scale study[J]. NeuroImage, 2017, 170: 456-470. |
| [13] | Cao X, Lin Y. Caggnet: crossing aggregation network for medical image segmentation[C]//2020 25th International Conference on Pattern Recognition(ICPR). Milan: IEEE, 2020: 1744-1750. |
| [14] | Isensee F, Jaeger P F, Kohl S A A, et al. U-Net: a self-configuring method for deep learning-based biomedical image segmentation[J]. Nature Methods, 2021, 18(2): 203-211. DOI:10.1038/s41592-020-01008-z |
| [15] | Chen L C, Papandreou G, Kokkinos I, et al. DeepLab: semantic image segmentation with deep convolutional nets, atrous convolution, and fully connected CRFs[J]. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2018, 40(4): 834-848. DOI:10.1109/TPAMI.2017.2699184 |
| [16] | Francesco V, Adriana R, Kyunghyun C, et al. ReSeg: a recurrent neural network-based model for semantic segmentation[C]// Proceedings of 29th IEEE Conference on Computer Vision and Pattern Recognition Workshops. Las Vegas, 2016: 426-433. |
| [17] | Sérgio P, Pinto A, Oliveira J, et al. Automatic brain tissue segmentation in MR images using random forests and conditional random fields[J]. Journal of Neuroscience Methods, 2016, 270: 111-123. DOI:10.1016/j.jneumeth.2016.06.017 |
| [18] | Rajchl M, Baxter J S, Mcleod A J, et al. Hierarchical max-flow segmentation framework for multi-atlas segmentation with Kohonen self-organizing map based Gaussian mixture modeling[J]. Medical Image Analysis, 2016, 27. |
| [19] | Andermatt S, Pezold S, Cattin P. Multi-dimensional gated recurrent units for the segmentation of biomedical 3D-data[M]. Cham: Springer International Publishing, 2016. https://doi.org/10.1007/978-3-319-46976-8_15. |
| [20] | Stollenga M F, Byeon W, Liwicki M, et al. Parallel multi-dimensional LSTM with application to fast biomedical volumetric image segmentation[C]//29th Annual Conference on Neural Information Processing Systems(NIPS). Montreal, 2015: 464-450. |
| [21] | Valverde S, Oliver A, Cabezas M, et al. Comparison of 10 brain tissue segmentation methods using revisited IBSR annotations[J]. Journal of Magnetic Resonance Imaging, 2015, 41(1): 93-101. DOI:10.1002/jmri.24517 |
