
 , 刘维宾1,2, 董昕阳3, 宋雨朦1,2
, 刘维宾1,2, 董昕阳3, 宋雨朦1,2 1. 东北大学 医学与生物信息工程学院, 辽宁 沈阳 110169;
2. 东北大学 教育部医学影像与智能分析工程中心, 辽宁 沈阳 110169;
3. 约克大学 计算机学院, 英国 约克郡 YO10 5DD
收稿日期:2021-09-06
基金项目:国家自然科学基金青年基金资助项目(61902058); 中央高校基本科研业务费专项资金资助项目(N2019002)。
作者简介:代茵(1982-), 女, 黑龙江齐齐哈尔人, 东北大学副教授。
摘要:为解决脑脊液病理图像中部分细胞膜较为模糊, 与图像背景难以区分的问题, 采用了基于注意力机制的U-Net深度学习方法对脑脊液病理图像做全自动分割.在深度学习网络中加入注意力机制对细胞进行定位, 抑制无关信息, 提高语义的特征表达, 提高对细胞整体分割的精确性.通过镜像、旋转等操作对数据集进行扩充预处理.采用VGG16预训练模型进行迁移学习, 交叉熵与Dice损失相结合作为损失函数, 分别在脑脊液临床图像与公开数据集2018 Data Science Bowl上进行验证;并与Otsu, PSPnet, Segnet, DeeplabV3+, U-Net进行对比, 结果表明, 本文方法在各项指标上均优于其他分割方法.
关键词:脑脊液检测细胞分割注意力机制深度学习U-Net模型
U-Net CSF Cells Segmentation Based on Attention Mechanism
DAI Yin1,2

 , LIU Wei-bin1,2, DONG Xin-yang3, SONG Yu-meng1,2
, LIU Wei-bin1,2, DONG Xin-yang3, SONG Yu-meng1,2 1. College of Medicine and Biological Information Engineering, Northeastern University, Shenyang 110169, China;
2. Engineering Center on Medical Imaging and Intelligent Analysis, Ministry of Education, Northeastern University, Shenyang 110169, China;
3. School of Computing, University of York, Yorkshire YO10 5DD, UK
Corresponding author: DAI Yin, E-mail: daiyin@bmie.neu.edu.cn.
Abstract: In order to solve the problem that part of the cell membrane in the pathological images of CSF (cerebrospinal fluid) is blurred and this is difficult to be distinguished from the image background. The U-Net based on attention mechanism is proposed to segment pathological images of CSF automatically. Attention mechanism is added to deep learning network to locate cells, suppress irrelevant information, improve semantic feature expression, and further improve the accuracy of cell segmentation. The datasets are preprocessed by mirroring and rotation. VGG16 pre-training model is used for transfer learning. Cross entropy is combined with Dice loss as Loss function which is validated in CSF clinical images and open dataset 2018 Data Science Bowl and compared with Otsu, PSPnet, Segnet, DeeplabV3+, U-Net. The results show that the proposed method is superior to other segmentation methods in all indexes.
Key words: CSF testcell segmentationattention mechanismdeep learningU-Net model
目前国内外针对自身免疫性脑炎的诊断研究是利用磁共振成像(magnetic resonance imaging, MRI)检测以及结合其他影像学诊断方法,如正电子发射计算机断层显像(positron emission computed tomography, PET)、超声波检查,以及脑脊液(cerebrospinal fluid, CSF)检测,而CSF检测是最为标准的,通常作为医生判断疾病的“金标准”[1].免疫机制在精神疾病病理学中的作用越来越得到人们的关注,多项研究表明了自身免疫性疾病与精神分裂症之间存在着必然的联系.CSF分析对识别中枢神经系统是否存在炎症起着核心的作用[2].CSF检测结果不仅可以作为判断脑炎与精神分裂症的依据,还可以作为临床孤立综合征[3]、额颞痴呆、小儿鞍上生殖细胞瘤、多发脑梗死性痴呆、神经莱姆病、遗传性多发脑梗死性痴呆、单发脑梗死性痴呆等疾病的重要依据.
CSF细胞图像有其独有的特点,其存在细胞边界模糊与背景不易区分、数据图像存在一定的噪声、相同类型的细胞通常在图像中成簇存在[4]、样本数据过少等问题.目前医生大多还是通过手动分割CSF细胞的方式并通过经验来诊断相关疾病,工作强度大,效率低,而基于人工智能的传统分割算法的鲁棒性差,所以针对CSF细胞检测的自动分割研究显得尤为重要.近几年深度学习发展迅猛,有研究人员在图像分割与深度学习相结合的领域作了许多研究与贡献.在CSF图像的研究中,Xu等[4]提出一种层次非最大抑制(hierarchy-non maximum suppression, Hierarchy-NMS)算法,与非极大抑制算法和软化非极大抑制算法进行了比较,证明了层次非最大抑制算法在细胞识别和计数方面均优于非极大抑制的一些变体.但该研究更多的关注点是识别与记数,而没有考虑CSF细胞边界与背景精确分割的问题.
在医学图像领域内,Jiang等[5]设计了一种基于双通道双向LSTM(dual channel bidirectional LSTM, DC-BLSTM)的神经网络,将3D图像分割任务转化为多个1D分割任务,比现有的基于卷积神经网络的3D神经元图像分割方法更容易标记训练样本.另外极具代表性的是以U-Net模型为基础框架,通过改造编码器或解码器实现其性能的提升.研究人员通过这些变体对许多细胞图像进行了分割研究,例如对腺体细胞进行分割[6],肌肉干细胞的分割[7],肺、细胞轮廓、胰腺的分割研究[8],多器官分割和心脏分割[9],以及对ISBI细胞追踪挑战赛2019数据集的分割测试[10]等.注意力机制的优秀性能与良好表现引起了研究人员的注意.Vaswani等[11]提出了Transformer网络架构,此架构是完全基于注意力机制,并且在WMT 2014 English-German数据集上验证了优越性能.Zhang等[12]提出一种带有注意力模块的O形网络结构,可以在不分割的情况下检测生物医学图像中的节点,使用该方法在两个视网膜数据集和一个神经元数据集中取得了良好的检测效果.迁移学习可以提升模型泛化能力,使模型的初始性能得到提高.刘珍梅[13]将迁移学习算法与宫颈细胞核分割算法相结合,减少分割标注数据集制作的压力,得到的模型收敛性更好.
本文将注意力机制与深度学习相结合,并引入迁移学习,对CSF细胞进行精确分割,取得了理想的结果.
1 方法描述本文采用了基于注意力机制的U-Net卷积神经网络对CSF细胞进行精确分割,并引入预训练模型进行迁移学习[14],在一定程度上提高了模型训练速度与性能.概括来说实验流程共分为4个部分:
1) 数据集扩充:将从已有的CSF图像中筛查出29例可靠图像,然后再采用对其旋转、裁剪、镜像的方法进行扩充;
2) 数据预处理:将扩充后的数据集进行尺寸大小统一预处理,使其适配batch的读取方式,网络的感受野也就限定了统一的尺度范围,方便统一做训练,并且训练集通过拉普拉斯算子进行了滤波处理;
3) 网络训练:将处理好的数据集传入带有注意力机制的U-Net卷积神经网络模型中,并引入迁移学习进行训练;
4) 分割指标评估:训练完成后,使用训练模型对数据进行预测,使其预测图与Ground Truth进行指标评估,评估指标包括相似系数值、平均交并比和类别平均像素准确率.
实验流程示意图如图 1所示.
图 1(Fig. 1)
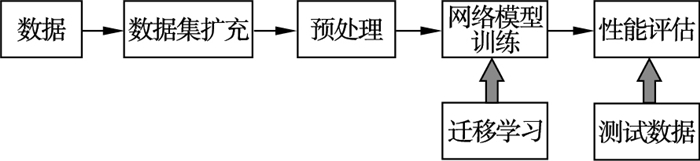 | 图 1 实验流程图Fig.1 Flow chart of the experiment |
2 基于注意力机制的U-Net网络结构模型2.1 总体设计本文所使用的基于注意力机制的U-Net卷积神经网络总体分为编码器和解码器,在解码器中加入注意力模块,在对编码器每个分辨率上的特征与解码器中对应特征进行拼接之前,使用Attention Gate重新调整了编码器的输出特征,该模块生成一个门控信号,用来控制不同空间位置处特征的重要性[15].网络的总体结构如图 2所示.
图 2(Fig. 2)
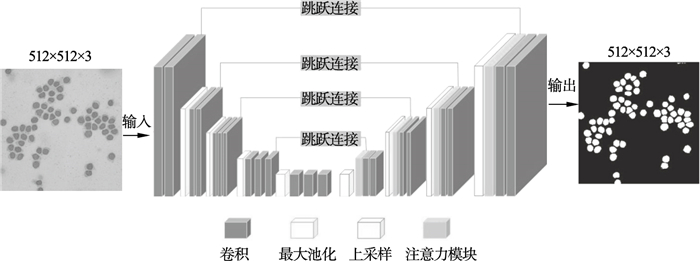 | 图 2 基于注意力机制的U-NetFig.2 Attention-based U-Net |
2.2 U-Net网络结构2.2.1 编码器结构作为主干特征提取网络,部分是由卷积层和最大池化层组合而成的模块,整体结构与VGG[16]极为相似,所以本文采用VGG16的主干特征提取网络,如此也方便使用预训练权重进行迁移学习.
在编码器中,共进行了5次卷积模块的操作,输入图像大小为512×512×3,前2个卷积模块操作分别为2次卷积核为3×3的64通道和128通道的卷积操作,获得的初步有效特征层再分别进行一次2×2的最大池化,获得相应的特征层.后3个卷积模板操作分别为3次卷积核为3×3的256通道、512通道和512通道的卷积操作用来获得初步有效特征层.第3个卷积模块与第4个卷积模块再分别进行一次2×2的最大池化,第5个卷积模板不再做最大池化操作.编码器的结构图如图 3所示.
图 3(Fig. 3)
 | 图 3 编码器的结构图Fig.3 Structure diagram of the encoder |
2.2.2 解码器结构经过编码器的主干特征提取获得5个有效特征层,对这5个特征层进行特征融合,所采用的融合方式是对特征层进行上采样并堆叠.为了方便搭建网络以及考虑到通用性,本文设计的U-Net与Ronneberger等所提出的U-Net[17]略有区别.本文采用上采样时先进行2倍的上采样,然后进行特征融合,这样做的优势是最终得到的特征层与输入图片大小相同.解码器的结构图如图 4所示.
图 4(Fig. 4)
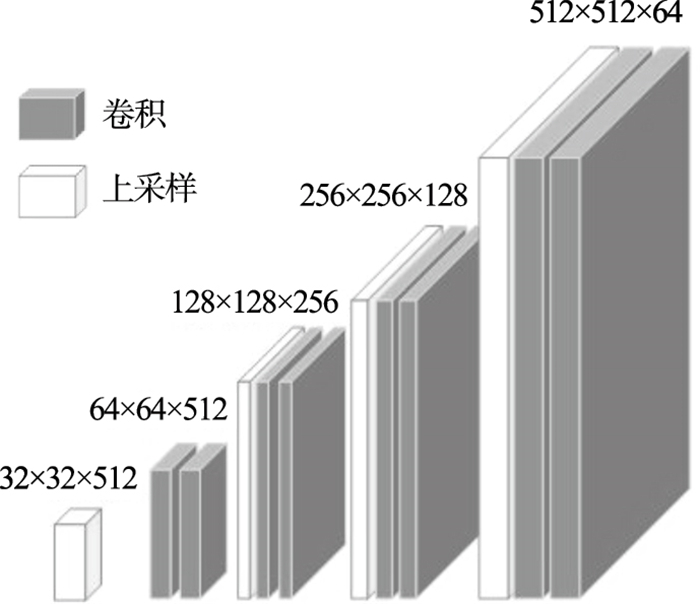 | 图 4 解码器的结构图Fig.4 Structure diagram of the decoder |
2.3 注意力机制在传统的U-Net中,为了避免在解码过程中丢失重要细节信息,使用了跳跃连接的方式,将编码过程中提取得到的映射直接链接到解码器相对应的层.但是这样操作有个缺点,即提取的低级特征会存在很多冗余信息.利用注意力机制[18]能够抑制模型学习与任务不相关的部分,强化学习与任务相关的特征,从原理上分为空间注意力与通道注意力.
Jaderberg等[19]提出的STN网络可以根据任务的不同,自适应地将数据进行空间变换.此网络是基于空间的注意力思想.SENet[20]在ILSVRC 2017分类中,赢得了第一名,此网络本质上是基于通道的注意力模型,它只使用全局平均池化来压缩通道特征.通过学习的方式自动获取每个特征通道的重要性,然后通过获取到的通道重要程度来提升有用信息和抑制无用信息.
本文采用的是通道注意力模块与空间注意力模块相结合的方式,将模块添加到网络的解码器中,通道的每一个高级特征都可以看作是一种特定的响应.通过挖掘通道映射之间的相互依赖关系,可以强调相互依赖特征映射,改善特定语义的特征表示.通道注意力模块与空间注意力模块相结合的模块示意图如图 5所示[15].
图 5(Fig. 5)
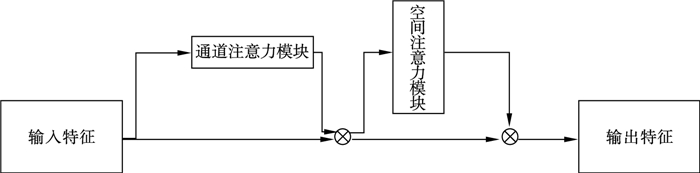 | 图 5 注意力模块Fig.5 Attention modules |
通道注意力专注于有意义的输入图像,为了有效地计算通道注意力,需要压缩输入特征图的空间维度.对于空间信息的聚合,常使用的方法是平均池化,而最大池化可以收集到难以区别物体之间的重要特征线索,来获取更加详细的通道注意力,所以通道注意力模块同时使用平均池化和最大池化.然后将它们送入一个权重共享的多层感知机中,最后再将这些输出的特征按对应位置相加.通道注意力模块如图 6所示[15].
图 6(Fig. 6)
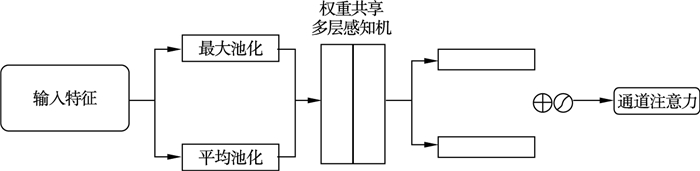 | 图 6 通道注意力模块Fig.6 Channel attention module |
空间注意力专注于输入图像的有效信息的位置,可以很好地弥补通道注意力的不足.为计算空间注意力,沿着通道轴使用平均池化和最大池化,将它们相连,生成一个有效特征描述符.最后通过卷积层生成空间注意力.空间注意力模块如图 7所示[15].
图 7(Fig. 7)
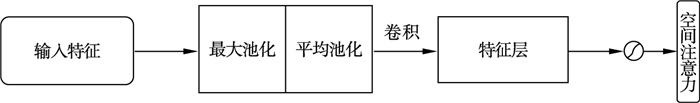 | 图 7 空间注意力模块Fig.7 Spatial attention module |
2.4 损失函数本文采用交叉熵与Dice损失相结合的形式作为语义分割的损失指标.交叉熵损失函数常用于分类问题,本文是对图像细胞做分割,本质也是对细胞与背景像素点做二分类,在二分类情况下,模型最后需要预测的结果有两种情况,对于每个类别预测出的概率为pi和1-pi,交叉熵的表达式为
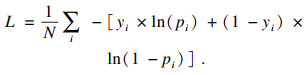 | (1) |
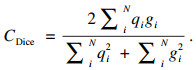 | (2) |
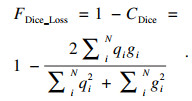 | (3) |
3 实验与分析3.1 数据及数据预处理本文采用的CSF图像信息数据集来自北京协和医院,采用HE染色技术获取,经过筛查得到29例可用数据作训练.这些训练数据集的标签是由专业人员手动标注而成,具有专业性.其标签的位深度为24 b,每个通道具有相同的像素数,且细胞像素设置为255,背景像素设置为0,所以标签的视觉效果是一个二值图像.
图像经过旋转、镜像、裁剪等方法将数据扩充至232例训练图像.并且将数据大小统一为512×512,以便训练.将扩充后的数据集经过一次拉普拉斯高通滤波处理,突出细胞边缘与背景的灰度差值,增强细节,其拉普拉斯核取为
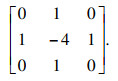 |
3.3 实验设置将扩充后的数据经过预处理后传入网络中作训练.网络参数的优化器使用Adam optimizer[22],此优化方法对高维度的数据进行快速拟合,利用梯度的一阶矩阵估计和二阶矩阵估计动态调整每个参数的学习率,经过偏置校正后,每次迭代学习率都有个确定范围,使得参数比较平稳.
为了均衡训练速度与训练效果,每次将2张图像传入网络中进行训练.加快网络训练速度,改善资源利用率,先将网络进行冻结,在冻结网络的情况下训练迭代50次,将更多的资源放在训练后面部分的网络参数.经过反复测试验证,冻结时的初始学习率设置为1×10-4效果最佳.迭代训练50次后,再将模型进行解冻,经过反复测试,这时的学习率设置为1×10-5效果最佳.模型解冻后继续迭代训练50次.当训练时,每隔3次当迭代模型性能不再提升时,则会采用每次将学习率降低原来学习率的0.5倍的方法来优化模型性能.
本文将实验数据按照8∶2的比例划分为训练集与验证集,将29例数据作为测试数据,来预测模型的实际分割效果.
3.4 评价指标与结果本文采用了语义分割中3个最常用的评价指标进行分析分割结果,它们分别为相似系数EDice,平均交并比U,以及类别平均像素准确率A.本文以二分类为例,计算公式为
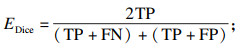 | (4) |
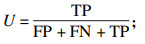 | (5) |
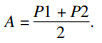 | (6) |
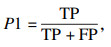 | (7) |
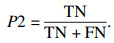 | (8) |
图 8(Fig. 8)
 | 图 8 细胞稀疏图像Fig.8 Sparse cell images (a)—原数据;(b)—金标准;(c)—Otsu;(d)—PSPnet;(e)—Segnet;(f)—DeeplabV3+;(g)—U-Net;(h)—本文方法. |
图 9(Fig. 9)
 | 图 9 细胞密集图像Fig.9 Dense cell images (a)—原数据;(b)—金标准;(c)—Otsu;(d)—PSPnet;(e)—Segnet;(f)—DeeplabV3+;(g)—U-Net;(h)—本文方法. |
如图所示,使用传统Otsu阈值分割时,属于背景的部分像素会被判定为细胞,对噪声极其敏感,对灰度差异不明显及不同目标灰度值有重叠的分割不明显.而PSPnet网络对医学图像的细节还原性较差,无法很好地识别医学图像的小目标物体.Segnet与DeeplabV3+模型分割尺寸相对差不多大小的细胞时,会存在细胞黏连的问题,并且分割尺寸相对较小且数量较多的细胞时,存在细胞分辨不清晰、分割模糊的问题.U-Net分割模型基本上实现了对所有细胞的有效分割,但是依然存在不足之处,该网络模型对细胞边界与背景之间的区分度略有不足.而使用本文的分割模型做预测分割时,上述所有问题均得到了改善.
使用上述分割模型分别对CSF细胞图像做出预测,EDice,U和A的具体结果如表 1所示.
表 1(Table 1)
| 表 1 CSF分割结果 Table 1 CSF segmentation results |
由表 1可看出,使用本文方法对CSF细胞图像做细胞分割要优于其他分割方法.
为了验证本文方法的普适性,选用2018 Data Science Bowl公开数据集作为参照实验数据.该数据包含大量分割的核图像,图像是在各种条件下获得的,细胞类型、放大倍数和成像方式各不相同.使用此数据得到预测分割的各项指标具体结果如表 2所示.
表 2(Table 2)
| 表 2 2018 Data Science Bowl数据集分割结果 Table 2 Segmentation results of 2018 Data Science Bowl dataset |
由表 2可看出,在2018 Data Science Bowl数据集分割中,本文方法要优于其他分割方法.
4 结论本文以CSF细胞为分割研究对象,采用基于注意力机制的U-Net网络,经过数据扩充与预处理,引入迁移学习,提高资源利用率,并且与Otsu,PSPnet,Segnet,DeeplabV3+以及U-Net做了对比实验,结果表明,本文方法要优于其他分割方法.并且通过公开数据集2018 Data Science Bowl做了进一步对比实验,实验结果进一步验证了本文分割方法在各项指标上均优于其他分割方法.
参考文献
| [1] | Graus F, Titulaer M J, Balu R, et al. A clinical approach to diagnosis of autoimmune encephalitis[J]. The Lancet Neurology, 2016, 15(4): 391-404. DOI:10.1016/S1474-4422(15)00401-9 |
| [2] | Kra B, Blf B, Hka B, et al. An observational study investigating cytokine levels in the cerebrospinal fluid of patients with schizophrenia spectrum disorders[J]. Schizophrenia Research, 2021, 231: 205-213. DOI:10.1016/j.schres.2021.03.022 |
| [3] | Tamam Y, Gunes B, Akbayir E, et al. CSF levels of HoxB3 and YKL-40 may predict conversion from clinically isolated syndrome to relapsing remitting multiple sclerosis[J]. Multiple Sclerosis and Related Disorders, 2021, 48: 102697. DOI:10.1016/j.msard.2020.102697 |
| [4] | Xu X, Li F, Wang S, et al. Hierarchy-NMS: merging candidate bounding boxes for cerebrospinal fluid cell image segmentation[J]. Journal of Physics: Conference Series, 2020, 1693: 012140-012144. DOI:10.1088/1742-6596/1693/1/012140 |
| [5] | Jiang Y, Chen W, Liu M, et al. 3D neuron microscopy image segmentation via the Ray-Shooting model and a DC-BLSTM network[J]. IEEE Transactions on Medical Imaging, 2020, 40(1): 26-37. |
| [6] | 赵宝奇, 尉飞, 孙军梅, 等. 结合密集连接块和自注意力机制的腺体细胞分割方法[J]. 计算机辅助设计与图形学学报, 2021, 33(7): 991-999. (Zhao Bao-qi, Wei Fei, Sun Jun-mei, et al. Glandular cell segmentation method combined with dense connective block and self-attention mechanism[J]. Journal of Computer-Aided Design & Computer Graphics, 2021, 33(7): 991-999.) |
| [7] | 张文秀, 朱振才, 张永合, 等. 基于残差块和注意力机制的细胞图像分割方法[J]. 光学学报, 2020, 40(17): 70-77. (Zhang Wen-xiu, Zhu Zhen-cai, Zhang Yong-he, et al. Cell image segmentation method based on residual block and attention mechanism[J]. Acta Optica Sinica, 2020, 40(17): 70-77.) |
| [8] | Ma H, Zou Y, Liu P X. MHSU-Net: a more versatile neural network for medical image segmentation[J]. Computer Methods and Programs in Biomedicine, 2021, 208: 106230-106239. DOI:10.1016/j.cmpb.2021.106230 |
| [9] | Chen J, Lu Y, Yu Q, et al. TransUNet: transformers make strong encoders for medical image segmentation[EB/OL]. (2021-02-08)[2021-11-21]. https://arxiv.org/abs/2102.04306v1. |
| [10] | Chan S X, Huang C, Bai C, et al. Res2-UNeXt: a novel deep learning framework for few-shot cell image segmentation[J]. Multimedia Tools and Applications, 2022, 81: 13275-13288. DOI:10.1007/s11042-021-10536-5 |
| [11] | Vaswani A, Shazeer N, Parmar N, et al. Attention is all you need[EB/OL]. (2017-12-06)[2021-12-23]. https://arxiv.org/abs/1706.03762v5. |
| [12] | Zhang Y, Liu M, Yu F, et al. An O-shape neural network with attention modules to detect junctions in biomedical images without segmentation[J]. IEEE Journal of Biomedical and Health Informatics, 2022, 26(2): 774-785. DOI:10.1109/JBHI.2021.3094187 |
| [13] | 刘珍梅. 基于BTTFA和迁移学习的宫颈细胞图像分割算法研究[D]. 广州: 华南理工大学, 2020. (Liu Zhen-mei. Nuclear segmentation based on BTTFA and transfer learning for cevicl cell images[D]. Guangzhou: South China University of Technology, 2020. ) |
| [14] | Weiss K, Khoshgoftaar T M, Wang D D. A survey of transfer learning[J]. Journal of Big Data, 2016, 3(1): 1-40. DOI:10.1186/s40537-015-0036-x |
| [15] | Woo S, Park J, Lee J Y, et al. CBAM: convolutional block attention module[C]//European Conference on Computer Vision. Berlin: Springer, 2018: 3-19. |
| [16] | Simonyan K, Zisserman A. Very deep convolutional networks for large-scale image recognition[EB/OL]. (2015-04-10)[2021-11-24]. https://arxiv.org/abs/1409.1556v1. |
| [17] | Ronneberger O, Fischer P, Brox T. U-Net: convolutional networks for biomedical image segmentation[C]//International Conference on Medical Image Computing & Computer-Assisted Intervention. Munich: Springer, 2015: 234-241. |
| [18] | Oktay O, Schlemper J, Folgoc L L, et al. Attention U-Net: learning where to look for the pancreas[EB/OL]. (2018-05-20)[2021-12-01]. https://arxiv.org/abs/1804.03999v1. |
| [19] | Jaderberg M, Simonyan K, Zisserman A. Spatial transformer networks[C]//Neural Information Processing Systems. Cambridge: MIT Press, 2015: 2017-2025. |
| [20] | Hu J, Li S, Gang S, et al. Squeeze-and-excitation networks[J]. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2020, 42(8): 2011-2023. |
| [21] | Ma J, Nie Z, Wang C, et al. Active contour regularized semi-supervised learning for COVID-19 CT infection segmentation with limited annotations[J]. Physics in Medicine and Biology, 2020, 65(22): 225034-225047. DOI:10.1088/1361-6560/abc04e |
| [22] | Kingma D P, Ba J. Adam: a method for stochastic optimization[EB/OL]. (2017-01-30)[2021-12-22]. https://arxiv.org/abs/1412.6980v2. |
