鉴于此,中国科学院沈阳应用生态研究所微生物资源与生态组团队在该领域进行了探索性研究,通过“自上而下”的合成生物学策略,从被氯嘧磺隆污染的土壤中富集驯化出可降解氯嘧磺隆的菌系L1,并通过宏基因组和16S rDNA测序技术对菌系在降解过程中进行了动态的微生物组学分析。
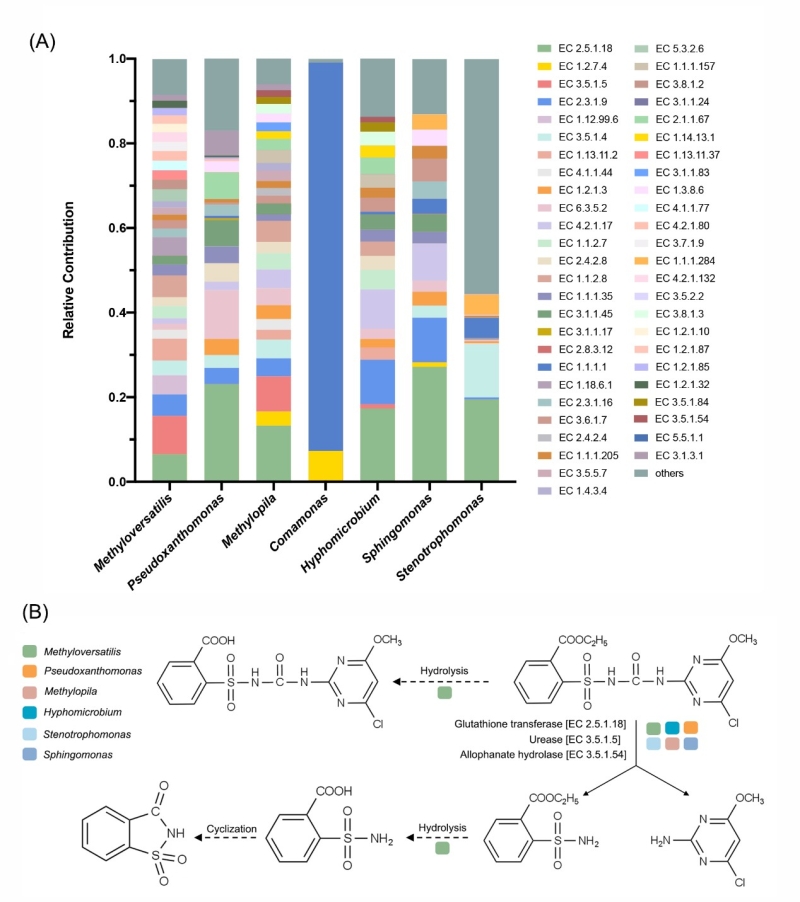
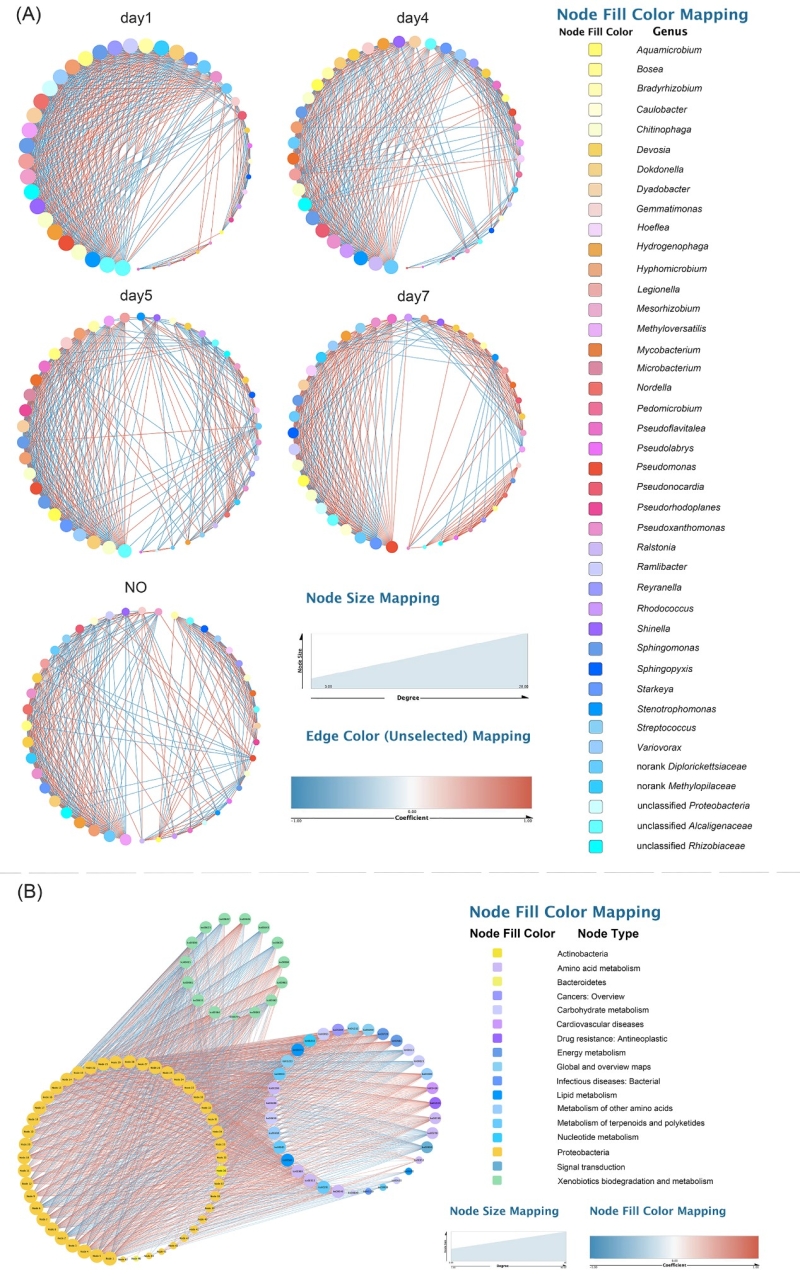
研究发现,该菌系在6天内可降解98.04%初始浓度为100mg L-1的氯嘧磺隆,其群落组成共有39个属,其中Methyloversatilis、Starkeya和Pseudoxanthomonas的丰度相对较高。菌系L1在不同时间点的群落组成未发生明显改变,但群落结构存在显著差异。基于KEGG的功能贡献度分析预测谷胱甘肽转移酶、脲酶和脲水解酶与氯嘧磺隆的降解相关,而Methyloversatilis, Pseudoxanthomonas, Methylopila, Hyphomicrobium, Stenotrophomonas, Sphingomonas是可能的主要降解菌属。菌系L1对氯嘧磺隆的代谢途径主要通过脲桥的断裂,生成邻甲酸乙酯苯磺酰胺基甲酸酯和邻磺酰胺苯甲酸乙酯。共线性及物种-功能网络分析结果均表明不同菌属间存在密切的互作、共养和共代谢关系,这种联系不仅存在于“功能菌”之间,也存在于“功能菌”和“辅助菌”之间。本研究不仅可解析菌系降解氯嘧磺隆除草剂的微生物学机理,还提供了潜在的降解菌资源,有助于磺酰脲类除草剂面源污染生物修复技术的创制和应用。
该成果以“Characterizing the microbial consortium L1 capable of efficiently degrading chlorimuron-ethyl via metagenome combining 16S rDNA sequencing”为题于2022年发表在Frontiers in microbiology(2区TOP,IF=5.64)。博士研究生李想为第一作者,张惠文研究员、徐明恺研究员为共同通讯作者,该研究得到了中国科学院战略性先导科技专项(A类)黑土专项、沈阳市科技局重大科技成果转化(双百项目)、国家自然科学基金、辽宁省博士科研启动资金项目的资助。