
在野生动物研究中,相比血液、肌肉等样品,粪便更易获得。粪便中含有大量细菌和少量动物肠道脱落的细胞,可以提供动物DNA。但粪便DNA在实际遗传分析中成功率和准确性都无法预估,结果参差不齐。特别是进入基因组时代,用粪便中提取的DNA进行动物全基因组测序,可用数据产量极少,限制了粪便的使用。
造成这一困难的主要原因是在粪便DNA中细菌的DNA数量庞大,干扰测序结果,因此降低细菌DNA的含量,使动物DNA得到富集是提高粪便DNA可用性的关键。尽管已经研发了多种富集方法,如从粪便中纯化动物细胞再提取DNA的提取前富集(Pre-extraction enrichment)、通过DNA或RNA-Bait杂交从总DNA中富集动物DNA的提取后富集(Post-extraction enrichment)和提取中富集(Peri-extractionenrichment)。但这些方法操作复杂、成本高昂、对粪便质量要求较为严格,特别是富集效率远远达不到要求,在实际应用中存在较大的局限性。
我校徐艳春团队与深圳华大生命研究院的兰天明团队共同开发了一种简单、有效、便宜的提取中富集方法,命名为Peri-extractionEnrichment by SDS,简称PEERS。该方法的原理如下图)。粪便中的细菌细胞被覆一层致密的硬质细胞壁,而动物细胞则是裸露的。因而,采用阴离子表面活性剂十二烷基硫酸钠(SDS),通过控制其浓度和作用时间,在室温下优先裂解动物细胞,使细菌的结构保持完整。通过简单的低速离心将细菌沉降序列,动物细胞的碎片、DNA等保留在上清中,将其分离并采用常规方法进行提取,所获的DNA中含有丰富的动物DNA和少量的细菌DNA。
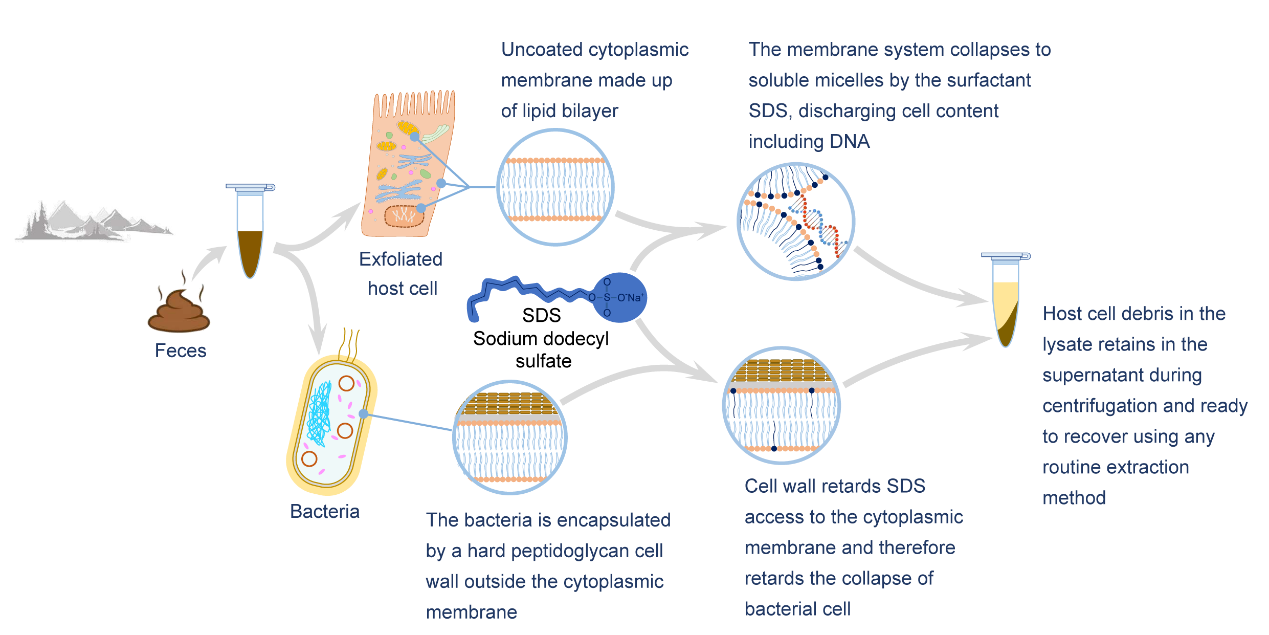
PEERS的工作原理
大量的测试表明,无论是肉食动物、草食动物还是杂食动物的粪便,PEERS对mtDNA和nuDNA的富集效率都明显高于当前的商业试剂盒,特别是在保存时间较长的粪便中更具优势。用PEERS富集的DNA进行微卫星和SNP两种传统分子标记的测试,当动物核DNA的拷贝数达到细菌DNA的1/1000时,SNP的基因型检测准确率可达90%或更高;当富集效率超过1/100时,SNP和和微卫星(STR)的准确性可与血液DNA相媲美。用PEERS富集的DNA进行全基因组重测序,当线粒体DNA富集到细菌DNA的1/10时(在绝大多数粪便中都很容易达到),就可以完美组装高质量的线粒体基因组。当核DNA富集到1/100时,即可实现对核基因组的全覆盖,测序质量与血液、肌肉等高质量DNA相当。
采用PEERS处理粪便每份样品的成本不到0.1美元,操作时间仅有几分钟,而且不增加任何额外的设备条件,极为简便。同时,这一技术还大大降低了获取野生动物遗传材料的难度,研究人员可以在不接触、看不见动物的情况下,获得动物的DNA开展遗传学和基因组学的研究。打破了基因库建设中依靠血液、肌肉、皮肤等损伤性材料的传统模式,使自然保护区巡护、疫源疫病监测、野生动物救护和动物园饲养等常态化工作中获取的粪便成为基因库的重要材料,大大拓展了样本来源空间。
