近年来,合成生物学作为一门新兴交叉学科获得了蓬勃发展,为破解人类面临的资源、健康、环境等重大挑战提供全新解决途径。合成生物学的核心理念是通过对DNA等生物大分子的逆向设计重构获得具有特定功能的人工生物系统。然而,这些生物分子编码的组合排列空间十分庞大,序列与功能的映射关系复杂,对生物大分子序列进行精准设计极具挑战。近期人工智能技术的突破引发了自然语言处理、计算机视觉等领域的革命性进步,尤其是以ChatGPT等为代表的生成式智能模型的突破,彰显出AI在提取复杂模式、生成复杂对象上的强大潜力。基于人工智能技术逆向设计启动子等具有特定功能的生物大分子序列,将为合成生物学的发展提供强大的设计工具和丰富的基础元件。
启动子是决定基因在何时、何地以何种程度进行转录表达的合成生物学基础元件,设计具有特定功能的人工启动子是逆向构造人工基因系统的基础。启动子中连接不同转录因子结合位点的之间的旁侧序列被证明对启动子功能有着重要影响,但这些旁侧序列的特征难以被人为归纳总结为明确的知识和设计准则。同时,由于天然基因组中具有特定转录因子结合序列的启动子数量稀少,难以直接建立深度学习模型对这些启动子的序列的整体模式进行提取。这些因素导致旁侧序列在设计中被长期忽视,缺乏有效的对启动子进行整体优化设计的方法。
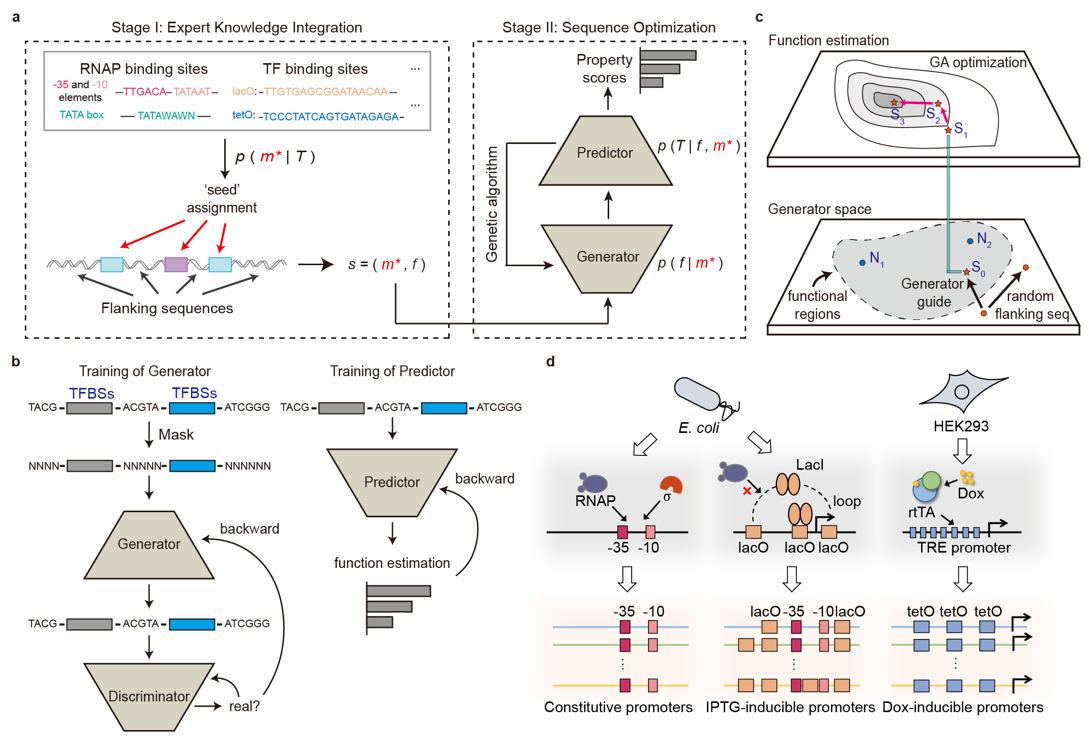
知识引导与数据驱动相融合的启动子辅助设计方法DeepSEED
针对这一问题,研究团队创新提出了一种知识引导与数据驱动相融合的智能设计策略:首先基于人类专家擅长在小样本中识别明确模式的特点,利用专家知识定义与启动子功能相关的重要显式模式作为“种子”序列;在此基础上,基于深度学习模型擅长在大型数据集中检测隐含弱模式的强大能力,在海量启动子数据中学习旁侧序列与种子序列的隐式匹配关系,进而基于条件生成式模型获得与特定种子序列相匹配的旁侧序列,对序列整体进行全局优化。在实际应用过程中,研究者可以任意指定已知生物调控模式序列作为“种子”序列,模型通过学习大数据中旁侧序列的调控规律对“种子”的旁侧序列进行补全,从而实现启动子的按需优化设计。研究团队成功将该方法应用于大肠杆菌内组成型启动子、IPTG诱导型启动子,以及哺乳动物细胞内Dox诱导型启动子的优化设计。模型设计生成的合成启动子在表现出高度序列多样性、与天然基因序列低相似性的同时,保留了天然序列中k-mer频率等关键统计特征,并优化了DNA序列大小沟偏好、偏转角等系统整体属性,大幅提升了合成启动子的转录活性和诱导率等关键性能。该成果有望为合成生物学研究提供基础性的设计工具和多样化的基因调控元件。
相关研究成果以“使用DeepSEED进行侧翼序列深度改造实现高效启动子设计”(Deep flanking sequence engineering for efficient promoter design using DeepSEED)为题,于10月9日发表于《自然·通讯》(Nature Communications)期刊。
清华大学自动化系博士研究生张鹏程、博士研究生王昊晨与硕士研究生许涵文为该论文的共同第一作者,汪小我教授为该论文的通讯作者。清华大学的魏磊、刘莉扬、胡志睿等也对本文作出了重要贡献。该研究得到国家自然科学基金、国家重点研发计划、清华大学国强研究院项目的资助。
论文链接:
https://www.nature.com/articles/s41467-023-41899-y
供稿:自动化系
题图设计:曾仪
编辑:李华山
审核:郭玲
2023年10月17日 11:13:13
