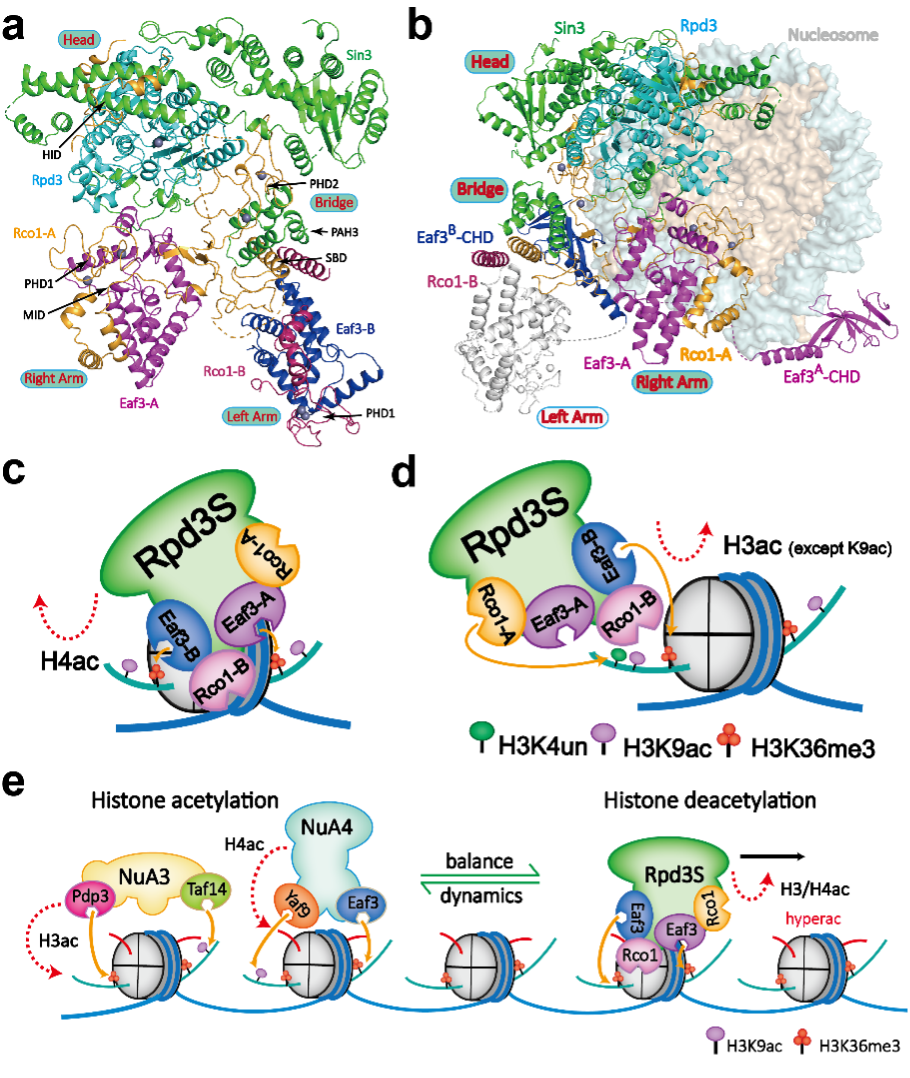
Rpd3S介导的染色质甲基化与乙酰化动态调控:a.组蛋白去乙酰化酶Rpd3S复合物的分子结构基础;b. Rpd3S复合物H3K36me3修饰引导下核小体催化与调控分子模型;c-d. Rpd3S在H3K4un和H3K36me3修饰引导下调控核小体组蛋白H4和H3去乙酰化过程机理;e. Rpd3S催化保留的H3K9ac与H3K36me3共同作为“种子”参与招募乙酰化转移酶NuA3/NuA4复合物促进乙酰化的快速重新建立。
近日,清华大学医学院李海涛教授课题组和生命学院闫创业副教授合作团队在Rpd3S介导的染色质去乙酰化动态调控机制上取得重要突破,研究成果以“组蛋白H3第36位赖氨酸三甲基化指引下的多模式Rpd3S核小体去乙酰化”(Diverse modes of H3K36me3-guided nucleosomal deacetylation by Rpd3S)为题,于7月19日在《自然》(Nature)期刊上作为研究长文发表。
首先,研究者利用化学生物学手段合成出性质均一的修饰组蛋白并成功组装出多种修饰组合类型的“人工设计”核小体。随后,通过单颗粒冷冻电镜技术解析了酿酒酵母Rpd3S复合物在自由状态和结合H3K36me3核小体状态下的分子结构模型。进而,研究者以复合多修饰核小体为底物系统探究了Rpd3S复合物去乙酰化酶活的特异性,并利用酵母遗传学等功能实验验证其调控模式。通过上述研究,研究者对Rpd3S复合物的组装模式、底物识别催化和修饰指引调控等过程进行了全面的分子机制解剖。本研究揭示了Rpd3S复合物在识别核小体底物和甲基化介导的去乙酰化调控过程中的动态和多样化模型,凸显出表观遗传调控的复杂精妙性,并展示了大自然通过形成多亚基大复合物来实现调控能力的精巧设计。
该研究成果展示Rpd3S的独特结构,其中两个Eaf3-Rco1异源二聚体与Rpd3和Sin3不对称地结合形成一个催化核心复合物。Eaf3、Sin3和Rco1分别参与对于两个H3K36me3修饰标签、核小体DNA和连接区DNA的多价识别,将Rpd3的催化中心定位在组蛋白H4氨基末端尾部附近进行去乙酰化酶活。在另一种催化模式中,Rco1和Eaf3通过识别非修饰的组蛋白H3第4位赖氨酸(H3K4un)和H3K36me3修饰从而引导特异性的组蛋白H3 N-末端尾部的去乙酰化酶活,却特异性保留了组蛋白H3第9位的乙酰化赖氨酸(H3K9ac)。依托分子结构模型、体外生化和体内功能数据,研究团队提出了H3K9ac和H3K36me3修饰可以作为“种子”修饰引导后续乙酰化转移酶的招募和快速H3/H4乙酰化修饰重新建立的机制。
李海涛和闫创业为文章的共同通讯作者,清华大学医学院博士后管海鹏和已毕业博士生王沛为并列第一作者。清华大学医学院2020级博士生张沛主要参与了该研究,上海交通大学医学院李兵教授、阮纯博士,以及郑州大学医学科学院郑向东****提供了专业指导和帮助。
该工作得到国家自然科学基金委员会、国家重点研发计划的资助。李海涛是清华-北大生命科学联合中心、北京生物结构前沿研究中心、分子肿瘤学全国重点实验室、教育部蛋白质科学重点实验室,以及山西医科大学-清华大学医学院前沿医学协同创新中心成员。
论文链接:
https://www.nature.com/articles/s41586-023-06349-1
科研成果动画展示:
https://static-content.springer.com/esm/art%3A10.1038%2Fs41586-023-06349-1/MediaObjects/41586_2023_6349_MOESM7_ESM.mp4
供稿:医学院
编辑:李若梦
审核:郭玲
2023年07月25日 13:27:11
