运用这种思路,研究团队报道了一种酶促组装具有重叠片段的DNA高级结构和双链片段的方法。研究的设计引入了一种具有3’至5’外切酶活性的DNA聚合酶(Q5 DNA聚合酶)来产生单链DNA粘末端,以实现特定的粘末端互补配对。通过精心的序列设计,Q5 DNA聚合酶的外切酶活性可以在只存在dTTP(不含有dATP、dCTP和dGTP)的情况下,消化预定序列3’末端只含有A,C和G核苷酸的部分。当酶作用到T核苷酸的位点时,由于体系中dTTP的存在,聚合酶活性会发挥作用,与外切酶活性进行一场“拔河”拉锯,从而使得消化作用被终止在特定位点。
图1.Q5 DNA聚合酶的聚合酶活性(左)和外切酶活性(右)的“拔河”拉锯
利用这种同一个酶中不同类型酶活性的对抗,研究实现了DNA纳米结构的自环化、同源多聚体以及包括二聚体、三聚体和四聚体在内的多种异源多聚体,复杂度最高达到了25个单元的一次性组装。研究还将此方法改进并应用回分子克隆领域,实现了“单一酶,一锅法”的体外DNA双链片段组装。
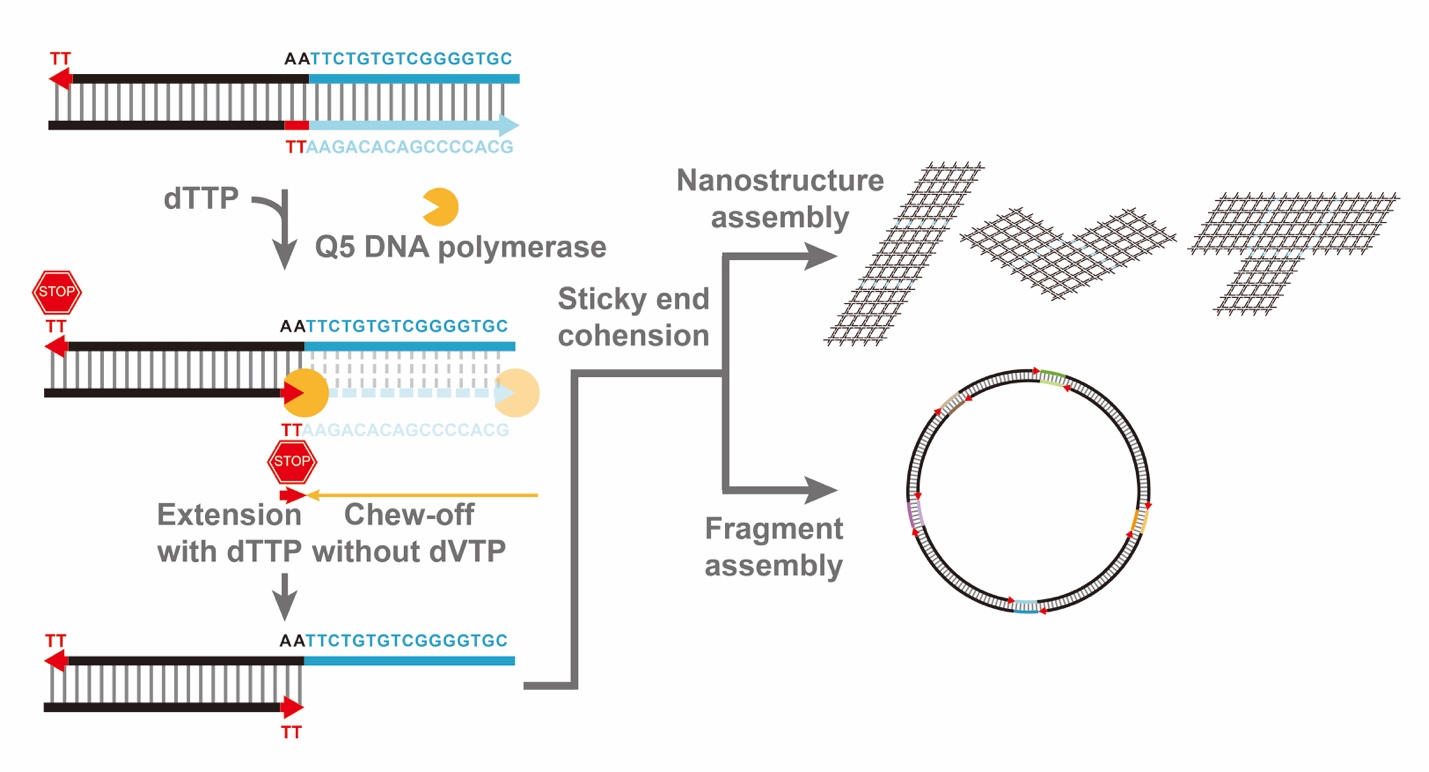
图2.DNA纳米结构和双链片段的组装原理
相关研究成果由清华大学生命科学学院魏迪明分子设计课题组(MADlab)和中国科学技术大学生命科学与医学部方芳课题组合作完成,论文题目为“具有重叠序列的DNA纳米结构和双链片段的酶促组装”(Enzymatic assembly of DNA nanostructures and fragments with sequence overlaps),于4月17日在线发表于《美国化学学会志》(Journal of the American Chemical Society)。
清华大学生命科学学院2020级博士生陈蓉为本文第一作者,清华大学生命科学学院魏迪明副教授和中国科学技术大学生命科学和医学部方芳教授为本文的共同通讯作者,清华大学生命科学学院已毕业的2018级本科生任洁灵和医学院医学实验班2018级本科生杨雨帆在清华大学生命科学学院本科生课程“核酸纳米结构的分子设计”中主导自主课程项目而为此研究作出了最初的探索。该研究得到科技部、国家自然科学基金委、清华-北大生命科学联合中心、清华大学春风基金、中国科学技术大学校创新团队培育项目、中国科学技术大学校青年创新重点基金项目等的资助。
论文链接:
https://doi.org/10.1021/jacs.3c01214
供稿:生命学院
题图设计:赵存存
编辑:李华山
审核:郭玲
2023年04月18日 16:00:48
