针对上述研究空白,清华大学深圳国际研究生院李炳副教授课题组利用多维组学((宏)基因组、宏转录组、蛋白组)结合微生物纯培养的方法,系统阐释了活性污泥菌群降解氯霉素的新机制。该研究通过宏基因组和基因组分析揭示了氯霉素降解菌群的群落结构和基因组特征;发现群落中的关键功能细菌首先将氯霉素的C1-OH和C3-OH基团氧化、C3异构化和C3-OH基团乙酰化,氯霉素的氧化产物、异构化产物和乙酰化产物经过一系列转化后裂解为对硝基苯甲酸(PNB)和2,2-二氯乙酸(DCA),最终这两种产物被群落中的其他成员完全矿化利用;Sphingomonas、Caballeronia、Cupriavidus三个关键菌属之间的协同合作实现了氯霉素的高度矿化和生物解毒。通过建立的多维组学分析方法解译了参与氯霉素代谢的关键酶,包括氯霉素氧化酶capO、乙酰基转移酶catB等。该研究为高浓度氯霉素废水(如制药废水、畜禽废水等)的强化生物处理提供了良好的菌株和酶资源。
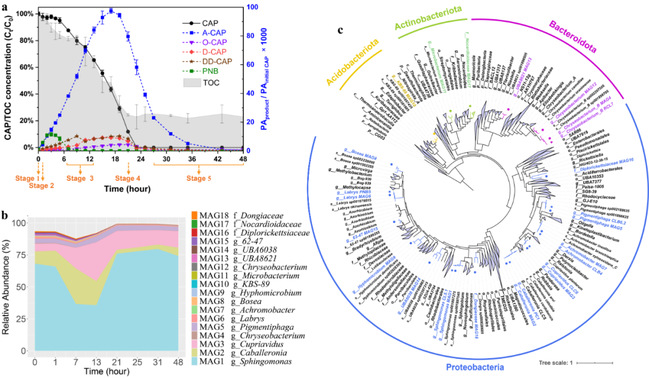
图1.氯霉素降解菌群的群落结构

图2.细菌体内氯霉素代谢途径与群落中微生物协同互作
相关研究成果以“应用多维组学和纯培养方法解译氯霉素的生物转化机制和微生物间相互作用”(Deciphering chloramphenicol biotransformation mechanisms and microbial interactions via integrated multi-omics and cultivation-dependent approaches)为题发表在微生物学领域国际期刊《微生物组》(Microbiome)。
本文通讯作者为清华大学深圳国际研究生院李炳副教授,第一作者为清华大学深圳国际研究生院博士后张家禹。本项目得到了国家重点研发计划(政府间国际科技创新合作)、国家自然科学基金委、广东省基础与应用基础研究基金、中国博士后基金的资助。
论文链接:
https://doi.org/10.1186/s40168-022-01361-5
供稿:深圳国际研究生院
编辑:李华山
审核:郭玲
2022年11月18日 11:20:50
